Chlamydien
| Chlamydiaceae | ||||||||
|---|---|---|---|---|---|---|---|---|
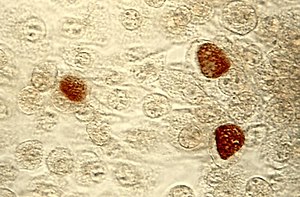
Chlamydia trachomatis, Einschlusskörperchen (braun) in einer McCoy-Zellkultur | ||||||||
| Systematik | ||||||||
| ||||||||
| Wissenschaftlicher Name | ||||||||
| Chlamydiaceae | ||||||||
| Rake 1957 (Approved Lists 1980) emend. Everett et al. 1999 | ||||||||
| Gattungen | ||||||||
|
Chlamydien ist eine in der Alltagssprache gebräuchliche Bezeichnung für Arten innerhalb der Familie Chlamydiaceae. Es handelt sich um sehr kleine, gramnegative Bakterien, die sich als Parasiten nur innerhalb einer Wirtszelle vermehren können. Sie können eine Vielzahl von Lebewesen infizieren, auch den Menschen. Eine Infektion mit Chlamydien wird als Chlamydiose bezeichnet. Die Erkrankungen betreffen u. a. die Schleimhäute im Augen-, Atemwegs- und Genitalbereich.
Die Vertreter der Chlamydiaceae durchlaufen in ihrem Entwicklungszyklus zwei Formen, als infektiöse Elementarkörperchen und als Retikularkörperchen, die die vegetative Form darstellen. Auf üblichen Nährmedien können sie nicht angezüchtet werden. Früher wurden alle bekannten Arten der Gattung Chlamydia zugerechnet, als Ergebnis genetischer Untersuchungen wurde 1999 die Systematik der Ordnung Chlamydiales, der Familie, der Gattungen und der Arten aktualisiert. Seitdem sind der Familie Chlamydiaceae zwei Gattungen – Chlamydia und Chlamydophila – zugehörig. Diese Systematik ist jedoch umstritten.
Merkmale
Erscheinungsbild
Die Vertreter der Chlamydiaceae können sich nur intrazellulär, d. h. innerhalb der Zellen eines Wirts vermehren. Die Chlamydien existieren in Form von stoffwechselinaktiven Elementarkörperchen (EK) – im Englischen elementary bodies, EB – von 0,2 bis 0,4 μm (Mikrometer) Durchmesser, die für den Infektionsweg bedeutsam sind. Nach einer Infektion finden sich 0,6–1,5 µm große Netzkörperchen (Retikularkörperchen, Abkürzung RK) in den Wirtszellen. Sie stellen die sich vermehrende, stoffwechselaktive Form dar.[1] In der englischen Fachliteratur werden sie als reticulate bodies (RB) bezeichnet, in der deutschen, v. a. medizinischen Literatur finden sich auch die Begriffe Initialkörperchen und Einschlusskörperchen.[2][3]
Die Vertreter der Chlamydiaceae werden als gramnegative Bakterien bezeichnet,[2][4] da sie – als Elementarkörperchen – in der Gramfärbung durch die verwendeten Farbstoffe rot angefärbt werden. Verursacht wird dies normalerweise durch eine dünne Mureinschicht (bestehend aus Peptidoglycanen) in der Zellwand, im Gegensatz zu den grampositiven Bakterien, die über eine dicke Mureinschicht verfügen. Genetische Untersuchungen haben bewiesen, dass die Vertreter der Familie Chlamydiaceae Gene besitzen, die für die Synthese der Peptidoglycane benötigt werden.[1] Allerdings zeigen die Untersuchungen, dass ihre Zellwände nur sehr geringe Mengen oder gar kein Peptidoglycan enthalten.[4]
Wachstum und Stoffwechsel
Chlamydien durchlaufen in ihrem Entwicklungszyklus zwei Formen: Außerhalb ihrer Wirtszellen existieren sie als Elementarkörperchen (EK). Das EK weist keine Stoffwechselaktivität auf, ist aber je nach Bakterienart durchaus resistent gegenüber Umwelteinflüssen (z. B. osmotischen Veränderungen und Austrocknung) und kann daher auch außerhalb des Wirtsorganismus überleben. Die Wirtszellen werden durch die Elementarkörperchen infiziert. Einmal in die Zelle aufgenommen, wandeln sich die Elementarkörperchen in Retikularkörperchen (RK) um, die einen aktiven Stoffwechsel besitzen und sich innerhalb der Wirtszelle vermehren.[1]
Das Elementarkörperchen muss in Kontakt mit der Wirtszelle treten, dabei handelt es sich um Epithelzellen. Es heftet sich an deren Membran[2] und wird von der Zelle in einer Art Beutel (ein als Endosom bezeichneter Membraneinschluss) aufgenommen, dieser Prozess wird als Endozytose bezeichnet. Die im Endosom eingeschlossenen EK wandeln sich innerhalb von ein bis zwei Stunden in die Retikularkörperchen (RK) um. Die Endosomen fusionieren nicht mit Lysosomen, wie dies eigentlich bei dem Prozess der Phagozytose passiert.[4] Forschungsergebnisse aus dem Jahr 2006 lassen vermuten, dass es den Retikularkörperchen gelingt, Lipide der Wirtszelle in ihre Hüllmembran einzubauen und somit zu verhindern, dass die Lysosomen der Wirtszelle sie als Fremdkörper erkennen und mit ihnen verschmelzen.[5] Da die RK nicht zerstört werden, vermehren sie sich innerhalb des Endosoms durch Zellteilung und sind dabei auf Stoffwechselprodukte und ATP (Adenosintriphosphat) der Wirtszelle für das Wachstum angewiesen. Die Generationszeit beträgt 2–3 Stunden.[1] Ihre Anhäufung im Endosom führt zu der Bezeichnung als Einschluss oder Einschlusskörperchen.[2][4] Anschließend verwandeln sie sich wieder in Elementarkörperchen zurück. Einige Tage nach der Infektion wird die Wirtszelle durch Lyse zerstört und die Elementarkörperchen werden dabei wieder freigesetzt, sie können nun weitere Zellen infizieren.[2] Es wurde auch beobachtet, dass die EK ohne Zerstörung der Zellen frei werden, indem die Endosomen mit der Zellmembran fusionieren und ihren Inhalt – die Elementarkörperchen – nach außen abgeben.[3]
Neben diesen beiden Hauptformen des Entwicklungszyklus kennt man noch eine weitere Form, die aberranten Körperchen. Dabei handelt es sich um eine intrazelluläre, persistierende Form, die unter nicht optimalen Wachstumsbedingungen der Wirtszellen entsteht. Sie wird als Dauerform mit reduziertem Stoffwechsel angesehen und kann sich wieder zu Elementarkörperchen entwickeln. Die aberranten Körperchen sind ebenfalls von medizinischer Bedeutung, da ihr Vorhandensein mit reaktiver Arthritis in Verbindung gebracht wird.[3]
Wegen des parasitären Stoffwechsels kann man Chlamydien nicht auf den in der Mikrobiologie üblicherweise verwendeten Nährmedien kultivieren, man benötigt stattdessen Zellkulturen.[4] Dabei werden üblicherweise HeLa-Zellen (menschliche Epithelzellen) verwendet,[6] auch die humanen Hep2- und die McCoy-Zelllinien werden eingesetzt.[3]
Die Vertreter der Chlamydiaceae besitzen – auf der Zellwand aufgelagert – Lipopolysaccharide. Diese Verbindungen aus fettähnlichen Bestandteilen, verbunden mit drei Zuckerbestandteilen, wirken als Antigen und können serologisch für den Nachweis verwendet werden.[2] Bei den in der Kernregion des Lipopolysaccharids vorhandenen Zuckerbausteinen handelt es sich um 2-Keto-3-desoxy-octonat (KDO), die drei Monosaccharide sind wie folgt mit einer glykosidischen Bindung verbunden: α-KDO-(2→8)-α-KDO-(2→4)α-KDO.[4]
Zur Unterscheidung von Chlamydia- und Chlamydophila-Arten ist die Erkenntnis von Bedeutung, dass nur erstere Glykogen als Speicherstoff produzieren können. Das Glykogen ist bei Chlamydia Arten in den Retikularkörperchen durch Iod-Kaliumiodid-Lösung (Lugolsche Lösung) nachweisbar und färbt diese Einschlusskörperchen braun an.[4]
Einige Stämme von Chlamydophila psittaci und Chlamydia trachomatis benötigen für das Wachstum ATP (Adenosintriphosphat), das ihnen durch die Wirtszelle geliefert wird, da sie es nicht selber synthetisieren können.[4] ATP ist der universelle Energieträger in jeder Zelle, daher wurden die Bakterien als Energieparasiten angesehen. Allerdings haben genetische Untersuchungen an einem Stamm von Chlamydia trachomatis gezeigt, dass dieser über Gene verfügt, die für die ATP-Synthese benötigt werden.[1] Zurzeit (2013) ist noch nicht abschließend geklärt, ob tatsächlich alle Vertreter der Chlamydiaceae auf ATP aus den Wirtszellen angewiesen sind.
Die osmotische Stabilität der Elementarkörperchen wird durch ein Hüllprotein aufrechterhalten. Es besteht aus mehreren Protein-Untereinheiten, die durch Disulfidbrücken verbunden sind. Ein Bestandteil wird als MOMP bezeichnet (Abkürzung für major outer-membrane protein (englisch), übersetzt etwa Hauptprotein der äußeren Membran). Dieses Protein weist eine Molekülmasse von etwa 40 kDa (Kilodalton) auf. Daneben kommen noch ein hydrophiles, Cystein-reiches Protein mit einer Molekülmasse von etwa 60 kDa und ein kleines Cystein-reiches Lipoprotein vor. Wenn das Elementarkörperchen eine Wirtszelle infiziert, werden die Disulfidbrücken innerhalb der Protein-Untereinheit und auch zwischen ihnen chemisch reduziert, ähnlich wie dies bei der Reduktion von Cystin geschieht. Dadurch wird die Umwandlung des Elementarkörperchens (EK) in das Retikularkörperchen (RK) ermöglicht.[4]
Genetik
Die Zellen weisen die Nukleinsäuren DNA und RNA auf, das ist von Bedeutung, da man bis etwa 1970 annahm, dass es sich bei diesen in der Medizin als Krankheitserreger bekannten Organismen um Viren handelt.[2] Das Genom mehrerer Bakterienstämme aus der Familie Chlamydiaceae wurde bereits vollständig sequenziert.[1] Die Größe des Genom aller bisher untersuchten Vertreter liegt zwischen 1000 und 1400 Kilobasenpaaren (kb),[4] das ist lediglich 25 % der Genomgröße von Escherichia coli. Die geringe Genomgröße ist ein Hinweis auf die parasitäre Lebensweise, die Chlamydien haben im Laufe der Zeit die Fähigkeit zur Synthese zahlreicher Metabolite verloren, da sie diese durch die Wirtszellen erhalten.
Das Ergebnis der Sequenzierung zeigt einen GC-Gehalt (den Anteil der Nukleinbasen Guanin und Cytosin) in der Bakterien-DNA von etwa 40 Mol-Prozent. Ein weiteres Kennzeichen ist, dass im Genom entweder ein oder zwei ribosomale Operons vorhanden sind. Ein Operon ist eigentlich eine Funktionseinheit der DNA, in diesem Fall hingegen ist das Operon in der ribosomalen RNA zu finden. Viele Vertreter (beispielsweise Chlamydophila felis) weisen neben dem Bakterienchromosom auch ein Plasmid mit einer Genomgröße von etwa 7,5 kb auf.[4]
Pathogenität
Die Vertreter der Chlamydiaceae sind alle als pathogen (krankmachend) eingestuft, da sie verschiedene Infektionskrankheiten verursachen. Mehrere Arten sind als Zoonoseerreger gekennzeichnet, da eine Infektion direkt oder indirekt zwischen Tieren und Menschen übertragen werden kann. Die genaue Zuordnung einer Art zu einer Risikogruppe erfolgt durch die Biostoffverordnung in Verbindung mit der TRBA (Technische Regeln für Biologische Arbeitsstoffe) 466.[7]
Nachweise
Der kulturelle Nachweis gilt als zuverlässige Methode zur Diagnostik von Chlamydien, wird aber nicht in der Routinediagnostik eingesetzt. Die Vertreter der Chlamydiaceae lassen sich nicht auf den in der Mikrobiologie üblicherweise verwendeten Nährmedien kultivieren, man benötigt stattdessen Zellkulturen. Dies wird vor allem von Speziallaboren durchgeführt, bei der Art Chlamydophila psittaci kommt noch die Schwierigkeit hinzu, dass die Anzucht nur in Laboren der Schutzstufe 3 zugelassen ist.[8] Der Nachweis erfolgt mit Hilfe der Fluoreszenzmikroskopie.[3]
Serologische Nachweise
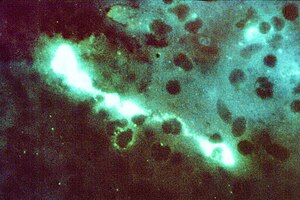
Die auf der Zellwand aufgelagerten Lipopolysaccharide wirken als Antigen und können serologisch im Rahmen der Antigen-Antikörper-Reaktion für den Nachweis verwendet werden. Häufiger erfolgt in der klinischen Diagnostik jedoch der Nachweis durch einen erhöhten Titer an Antikörpern, das dabei verwendete Antigen ist meist nicht speziesspezifisch und zeigt folglich mehrere Chlamydophila oder Chlamydia Arten an.[2] Weiterhin lässt sich durch den Nachweis der Antikörper nicht eindeutig zwischen ausgeheilten und persistierenden Infektionen unterscheiden.[9]
Der Nachweis spezifischer Antikörper im Serum erfolgt im Routinelabor bei Chlamydophila pneumoniae und C. psittaci auch mit dem ELISA-Verfahren. Auch hier gilt die Einschränkung, dass meist nur gattungsspezifische Antikörper nachgewiesen werden.[8]
Weitere serologische Verfahren vergleichen die Reaktivität der Antikörper im Patientenserum mit Antigenen von Chlamydia trachomatis, Chlamydophila pneumoniae und Chlamydophila psittaci. Das Antigen mit der stärksten Reaktivität zeigt die Spezifität der Antikörper an. Darauf basiert der Mikroimmunfluoreszenz-Test (MIF) und ein Immunblot (Western Blot). Für diesen werden rekombinante (gentechnisch hergestellte) Antigene benötigt. Für beide Verfahren gibt es kommerzielle Angebote.[9]
Häufig eingesetzt wird die Immunfluoreszenz. Sie basiert auf dem Antigen-Nachweis, der eingesetzte Antikörper ist mit einem fluoreszierenden Farbstoff markiert. Als Untersuchungsmaterial wird ein Abstrich verwendet, der die Bakterien enthält. Auch hier können durch nicht speziesspezifische Antikörper falsch positive Ergebnisse auftreten, die mit einem zweiten Verfahren bestätigt werden sollten. Sowohl die niedrige Sensitivität als auch die geringe Spezifität dieser Verfahren erlaubt den Chlamydiennachweis nur bei hoher Bakteriendichte. Ebenfalls auf diesem Prinzip basierende Schnelltests liefern keine zuverlässigen Ergebnisse.[9]
Antigen-Nachweisverfahren (neben der direkten Immunfluoreszenz auch das ELISA-Verfahren) werden trotz der geringen Spezifität häufig in der Veterinärmedizin eingesetzt – beispielsweise beim Nachweis von Chlamydophila psittaci –, da bei einer vorliegenden Infektion meist viele Bakterien in den Proben enthalten sind. Für die Diagnostik beim Menschen sind diese Verfahren nicht validiert und deshalb nicht zu empfehlen.[8]
Molekularbiologische Nachweise
Wesentlich spezifischer ist der Nachweis bestimmter Teile des bakteriellen Genoms mit Hilfe des PCR-Verfahrens (Polymerase-Kettenreaktion). Dabei werden Genabschnitte, die typisch für die Bakterienart sind, vervielfältigt (amplifiziert) und nachgewiesen.[2]
Im Rahmen der Diagnostik werden darauf basierende, sogenannte Nukleinsäure-Amplifikations-Techniken (NAT) eingesetzt, die eine hohe Sensitivität und Spezifität aufweisen. So wird beim Nachweis von C. trachomatis eine DNA-Sequenz aus dem Plasmid amplifiziert. Da das Plasmid in 10-facher Kopie in einer Zelle vorkommt, wird so eine hohe Sensitivität erreicht. Allerdings muss regelmäßig überprüft werden, ob die Zielsequenz tatsächlich noch in dem nachzuweisenden Bakterium vorkommt, sonst kommt es zu falsch negativen NAT-Resultaten. Zu diesem Zweck wird ein anderes NAT-Protokoll verwendet, also ein Verfahren mit einer anderen für die Art typischen Zielsequenz. Für C. trachomatis eignet sich hierfür das MOMP-Gen. Da die Nukleinsäure-Amplifikations-Technik eine hohe Sensitivität aufweist, können direkt klinische Proben, wie Abstriche, respiratorischen Proben oder Urin damit untersucht werden.[9]
Für den Nachweis von C. trachomatis hat die Weltgesundheitsorganisation (WHO) die bekannten Daten über verschiedene Untersuchungsverfahren ausgewertet: Dabei wurden als Proben Urin oder Genitalflüssigkeit verwendet. Dabei zeigen die Nukleinsäure-Amplifikations-Techniken mit etwa 87 % Sensitivität und 99,2–99,8 % Spezifität die zuverlässigsten Ergebnisse.[10]
Für den Nachweis von C. psittaci und C. pneumoniae steht bislang kein kommerzielles PCR-Protokoll zur Verfügung, so dass dieses Verfahren Speziallaboren vorbehalten ist.[8] Ein 2010 entwickeltes Verfahren beruht auf der Real Time Quantitative PCR (q-PCR), dabei wird der Fluoreszenzfarbstoff SYBR Green eingesetzt, der sich an die nachzuweisenden Genabschnitte anlagert und eine Fluoreszenz verursacht. Die Stärke der Fluoreszenz wird während eines PCR-Zyklus in Echtzeit erfasst (daher die Bezeichnung real time) und dient der quantitativen Bestimmung der vorhandenen Genabschnitte und somit einer quantitativen Erfassung der Bakterienart. Das in Japan entwickelte Verfahren zielt auf den Nachweis von C. psittaci ab, es lassen sich aber auch andere tierpathogene Chlamydophila Arten, u. a. C. felis, damit nachweisen. Es können klinische Proben (z. B. Faeces) oder infizierte Zellen eingesetzt werden, aus denen zunächst die Extraktion der DNA erfolgt, mit der dann die q-PCR durchgeführt wird.[6]
Vorkommen
Vorkommen von Chlamydia
Chlamydia trachomatis kommt ausschließlich beim Menschen als Krankheitserreger vor.[9] C. muridarum (vor 1999 als Variante von C. trachomatis angesehen) kommt bei Vertretern der Muridae (Mäuseartige) vor, beispielsweise bei Mäusen und Hamstern. Chlamydia suis ist bei Schweinen zu finden, der natürliche Wirt ist das Wildschwein, sie kommt aber ebenso beim Hausschwein vor. C. felis (ehemals Chlamydophila felis) ist endemisch bei Hauskatzen, der Erreger ist weltweit nachweisbar. Chlamydia abortus (ehemals Chlamydophila abortus) ist endemisch bei Wiederkäuern, wie Rindern, Schafen und Ziegen. In einzelnen Fällen wurde sie auch bei Pferden, Kaninchen, Meerschweinchen und Mäusen nachgewiesen, durch eine Zoonose ebenfalls in Einzelfällen beim Menschen.[4]
Vorkommen von Chlamydophila
Chlamydophila pneumoniae kommt nahezu ausschließlich beim Menschen vor, der Erreger ist weltweit nachweisbar. Vereinzelt wurden Biovare bei Tieren gefunden, dies waren Pferde und Koalas, ein Zusammenhang mit menschlichen Erkrankungen konnte bisher nicht gezeigt werden.[8] Das natürliche Reservoir von C. psittaci sind Vögel, besonders Papageien. 2012 erfolgte eine genetische Untersuchung der bekannten Genotypen von Chlamydophila psittaci, diese wurden aus folgenden Vögeln isoliert: Kakadus, Wellensittiche, Loris und anderen Papageien, Tauben, Enten, Gänse, Truthühner und Laufvögel. Einige Stämme wurden auch bei Menschen (als Folge der Ornithose), Hausrindern und Bisamratten gefunden.[11]
C. pecorum lässt sich bei verschiedenen Säugetieren finden, ohne dass bisher ein spezifischer Wirt erkannt wurde. Gefunden wurde sie bei Wiederkäuern (Rindern, Schafen und Ziegen), Koalas und Schweinen. C. caviae kommt beim Meerschweinchen vor, insbesondere bei Vertretern der Gattung Cavia (echte Meerschweinchen), zu denen auch das Hausmeerschweinchen gehört. Es wird angenommen, dass sie spezifisch für diese Gattung ist, denn Versuche andere verwandte Nagetiere (u. a. Hamster und Rennmäuse) zu infizieren, schlugen – mit einer Ausnahme – fehl.[4]
Vorkommen von weiteren Vertretern der Ordnung
Die zur Ordnung Chlamydiales gehörende Art Simkania negevensis kommt ebenfalls als Krankheitserreger beim Menschen vor, sie wurde aber auch in frei lebenden Amöben nachgewiesen.[8] Für die ebenfalls dieser Ordnung angehörende Familie der Parachlamydiaceae ist dies typisch, sie sind fast ausschließlich in Amöben zu finden.[4]
Systematik
Der Name Chlamydien leitet sich aus dem Griechischen her, χλαμύς (chlamys, Genitiv chlamydos) bezeichnet einen kurzen Mantel, siehe auch „Chlamys (Mantel)“.[12]
Aufgrund ihrer geringen Größe und ihrer rein intrazellulären Vermehrung rechnete man Chlamydien bis in die 1960er Jahre den Viren zu. 1966 wurden sie als eigene Ordnung Chlamydiales der Bakterien erkannt. Aufgrund von neueren 16S rRNA-Analysen werden sie nun zu der Abteilung Chlamydiae gestellt. Weitere Untersuchungen haben eine enge Verwandtschaft der Chlamydiae und den Abteilungen der Verrucomicrobia und Planctomycetes gezeigt. Aufgrund dieser Untersuchungen bilden diese drei Abteilungen den sogenannten PVC-Superphylum.[13]
Nach der aktuellen Systematik ist die Ordnung Chlamydiales in vier Familien aufgeteilt:[12]
- Chlamydiaceae Rake 1957
- Parachlamydiaceae Everett et al. 1999
- Simkaniaceae Everett et al. 1999
- Waddliaceae Rurangirwa et al. 1999
Vor 1999 waren alle bekannten Vertreter der Familie Chlamydiaceae zugeordnet, die im medizinischen Bereich als Chlamydien bezeichnet wurden, das gemeinsame Merkmal war die parasitäre Lebensweise.
Diese morphologisch orientierte Systematik in der Mikrobiologie wird gestützt durch chemotaxonomische Merkmale und biochemische bzw. stoffwechselphysiologische Eigenschaften. Seit genetische Untersuchungen durch Sequenzierung der DNA (Desoxyribonukleinsäure) und RNA (Ribonukleinsäure) möglich sind, benutzt man diese zur Aufklärung der Stammesgeschichte und der verwandtschaftlichen Beziehungen der Organismen untereinander. Dabei wird neben den DNA-Sequenzen des Genoms vor allem bei Bakterien zusätzlich die 16S rRNA untersucht, ein für Prokaryoten typischer Vertreter der ribosomalen RNA. Genetische Untersuchungen der DNA-Sequenzen erfolgen z. B. mittels DNA-DNA-Hybridisierung.[1]
Durch die Untersuchungen von Everett u. a. erhielt man ein genaueres Bild über die vorliegenden Verwandtschaftsverhältnisse bei den Chlamydien. Dabei wurden unter anderem die Sequenzen der 16S und 23S rRNA untersucht und mit bereits vorhandene Daten im Rahmen einer phylogenetischen Analyse verglichen, mit denen dann ein phylogenetischer Baum erstellt werden kann. Außerdem wurden Kriterien aufgestellt, mit denen man bei taxonomisch noch nicht eindeutig festgelegten Organismen prüfen kann, ob diese Vertreter der Familie Chlamydiaceae sind. Dazu wird der Stamm B577 quasi als typisches Beispiel der Familie definiert. Ein Isolat muss mehr als 90 % Übereinstimmung mit den 16S rRNA Genen von Stamm B577 aufweisen, um als Vertreter der Familie anerkannt zu werden.[4]
Die Ergebnisse von Everett, Andersen und Bush – vor allem die dadurch entstandene neue Systematik – wurden von der Fachwelt kritisch bewertet. Neben Zweifeln an den Kriterien zur taxonomischen Einstufung wurde 2001 hauptsächlich die Sorge geäußert, dass die nun neuen Bezeichnungen Chlamydia und Chlamydophila bei Angehörigen des Gesundheitswesens zur Verwirrung führen könnten.[14] Hintergrund ist, dass die Erreger weltweit Krankheiten verursachen und daher der Begriff der Chlamydia bzw. Chlamydien nur über einen längeren Zeitraum zu etablieren war. Die neue Taxonomie gilt nach den Kriterien der Systematik der Bakterien jedoch als valide publiziert.[12] Everett und Andersen verwiesen in einem Antwortschreiben an die Kritiker darauf, dass auch bedeutende Sequenzdatenbanken (wie GenBank) und Sammlungen von Mikroorganismen (wie die ATCC) die Bezeichnungen bereits übernommen haben.[14] Seitdem wird die Bezeichnung Chlamydien (bzw. chlamydiae im Englischen) vor allem im medizinischen Bereich sowohl für die Familie Chlamydiaceae als auch für weitere Vertreter in der Ordnung Chlamydiales verwendet,[3] zum Teil findet sich auch in der mikrobiologischen Fachliteratur immer noch die veraltete Gattungsbezeichnung Chlamydia für Spezies der Gattung Chlamydophila.[11]
2009 wurde von einigen Wissenschaftlern vorgeschlagen, wieder die frühere Gattungsbezeichnung Chlamydia bei allen Arten zu verwenden. Als Begründung wurde angeführt, dass die Ergebnisse der 16S rRNA Sequenzanalysen nicht mit der natürlichen Evolutionsgeschichte vereinbar seien.[15] Bisher (Stand 2018) sind nach dem Internationalen Code der Nomenklatur der Bakterien immer noch beide Gattungsbezeichnungen – Chlamydophila und Chlamydia – gültige Taxa, denen entsprechend der von Everett u. a. neu eingeführten Systematik die bekannten Arten zugeordnet werden, es erfolgten allerdings bereits Reklassifizierungen von einigen Chlamydophila- als Chlamydia-Arten.[16] 2009 wurde von der Internationalen Kommission für die Systematik der Prokaryoten ein Unterausschuss (Subcommittee on the taxonomy of the Chlamydiae, „Unterausschuss für die Taxonomie der Chlamydien“) gegründet, der sich mit diesem kontroversen Thema befasst.[17] 2010 wurde von diesem Unterausschuss eine Arbeitsgruppe eingerichtet. Ihre Aufgabe ist es, alle Daten zu prüfen, die sich aus den bisher vorhandenen Genomsequenzierungen ergeben, um festzustellen, ob die genetischen Abweichungen ausreichend sind für die Etablierung von zwei Gattungen.[18] Bezüglich der Kriterien für die Zugehörigkeit zur Familie Chlamydiaceae wurde 2011 von dem Unterausschuss bestätigt, dass die phylogenetische Untersuchung der Sequenzen der 16S und 23S rRNA geeignet ist. Darüber hinaus sollen auch weitere Gene (englisch core genes, „Kern-Gene“) geprüft werden, die eher Unterschiede erkennen lassen als die Gene der ribosomalen RNA.[19] Ein weiterer Vorstoß erfolgte 2015 durch eine Gruppe von Wissenschaftlern, die erneut anzweifelten, dass die phylogenetischen Untersuchungen die Aufteilung auf zwei Arten rechtfertigen.[16]
Das folgende Kladogramm stellt eine Hypothese der Verwandtschaftsverhältnisse innerhalb der Ordnung Chlamydiales dar (Stand 2001):[20]
| N.N. |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Zur Familie Chlamydiaceae gehören die beiden Gattungen Chlamydia und Chlamydophila, die zahlreiche human- und tierpathogene Arten enthalten. Chlamydia trachomatis bzw. Chlamydophila psittaci sind jeweils die Typusarten. Aktuell (Stand 2018) sind die folgenden Arten der beiden Gattungen bekannt:[12]
- Chlamydia Jones et al. 1945 (Approved Lists 1980) emend. Everett et al. 1999
- Chlamydia abortus (Everett et al. 1999) Sachse et al. 2015, comb. nov. (vorher Chlamydophila abortus Everett et al. 1999, sp. nov. [als Basonym], davor eine Variante von Chlamydia psittaci)
- Chlamydia avium Sachse et al. 2015, sp. nov.
- Chlamydia felis (Everett et al. 1999) Sachse et al. 2015, comb. nov. (vorher Chlamydophila felis Everett et al. 1999, sp. nov. [als Basonym], davor eine Variante von Chlamydia psittaci)
- Chlamydia gallinacea Sachse et al. 2015, sp. nov.
- Chlamydia muridarum Everett et al. 1999 (vorher eine Variante von Chlamydia trachomatis)
- Chlamydia suis Everett et al. 1999 (vorher eine Variante von Chlamydia trachomatis)
- Chlamydia trachomatis (Busacca 1935) Rake 1957 emend. Everett et al. 1999.
- Chlamydophila Everett et al. 1999, gen. nov.
- Chlamydophila caviae Everett et al. 1999, sp. nov. (vorher eine Variante von Chlamydia psittaci)
- Chlamydophila pecorum (Fukushi & Hirai1992) Everett et al. 1999, comb. nov. (vorher Chlamydia pecorum)
- Chlamydophila pneumoniae (Grayston et al. 1989) Everett et al. 1999, comb. nov. (vorher Chlamydia pneumoniae)
- Chlamydophila psittaci (Lillie 1930) Everett et al. 1999, comb. nov. (vorher Chlamydia psittaci)
Medizinische Bedeutung
Unbehandelt kann eine Chlamydiose der Geschlechtsorgane zu einer Unfruchtbarkeit führen.[21] Eine Infektion der Eileiter (Tuba) führt häufig zu Vernarbungen und Verwachsungen und kann so Funktionseinschränkung verursachen. Chlamydieninfekte sind eine der Hauptursachen für die tubare Unfruchtbarkeit von Frauen. Auch bei Männern können aufsteigende Urogenitalinfekte zu späteren Fruchtbarkeitsstörungen führen.[22]
Überblick über pathogene Arten
Alle bekannten Arten der Familie sind Krankheitserreger bei Mensch und Tier. Eine Infektion mit Chlamydien wird als Chlamydiose bezeichnet, je nach beteiligter Art oder sogar Serotyp gibt es unterschiedliche Krankheiten, hier soll nur ein Überblick gegeben werden.
| Arten bzw. Serovare | Krankheiten bzw. Informationen |
|---|---|
| Chlamydia trachomatis | C. trachomatis hat mehrere Untergruppen, die sich durch ihre Oberflächenstruktur unterscheiden und so in Serovare (Serotypen) eingeteilt werden. Diese Serovare wiederum lassen sich in drei Gruppen anordnen, die unterschiedliche Erkrankungen verursachen. |
| C. trachomatis Serovar A bis C |
Serovare A bis C sind die Erreger des Trachoms, einer schwerwiegend verlaufenen Form der Bindehautentzündung (Konjunktivitis). Es ist eine der häufigsten Augenerkrankung in Entwicklungsländern, sie führt dort oft zur Erblindung. In Europa ist das Trachom dagegen selten. Die Übertragung erfolgt vor allem durch mangelnde Hygiene. |
| C. trachomatis Serovar D bis K |
Serovare D bis K sind die in den Industrieländern am häufigsten vorkommende Untergruppe und können Infektionen im Genitalbereich auslösen, die oft vereinfacht nur als Chlamydiose bezeichnet werden. Dabei handelt es sich um eine sexuell übertragbare Krankheit. Auch die durch Chlamydien verursachte Konjunktivitis und Pneumonie bei Neugeborenen sowie das Paratrachom (Schwimmbadkonjunktivitis) bei Erwachsenen werden durch diese Serovare verursacht. Als Spätfolge einer Infektion kann in seltenen Fällen bei Männern eine reaktive Arthritis (im Zusammenhang mit Morbus Reiter) auftreten.[3] |
| C. trachomatis Serovar L1 bis L3 |
Serovare L1 bis L3 lösen das Lymphogranuloma venereum aus, das Ursache von ulcerösen Proktitiden, wie auch schmerzhaften Lymphknotenschwellungen in den Leisten, einer Urethritis oder auch einer Epididymitis sein kann. |
| Chlamydophila pneumoniae | C. pneumoniae, die viel weiter verbreitet als C. trachomatis ist, ruft hauptsächlich Lungenentzündungen (Pneumonien) hervor. Weiterhin kann sie unter anderem chronischen Husten auslösen und steht auch im Verdacht, bei Herzerkrankungen aufgrund von Arteriosklerose eine Rolle zu spielen. |
| Chlamydophila psittaci |
C. psittaci ist der Erreger der Ornithose (Papageienkrankheit). Das natürliche Reservoir von C. psittaci sind Vögel, besonders Papageien, die selber von der Infektion betroffen sein können. Als Zoonose ist sie auf den Menschen übertragbar. Die Infektionskette endet dort in der Regel. Vögel, u. a. von Tierfarmen, aber auch Papageien und Tauben spielen als Infektionsquelle für den Menschen die wichtigste Rolle. Diese Erkrankung ist beim Menschen sehr selten und ähnelt einer schweren Lungenentzündung.[8] |
| Chlamydia felis (Chlamydophila felis) | C. felis ist, neben Bordetella bronchiseptica, ein bakterieller Erreger des Katzenschnupfens. Die durch C. felis verursachte Infektionskrankheit wird auch als Feline Chlamydiose bezeichnet, sie wird in Einzelfällen auch auf den Menschen übertragen.[4] |
| Chlamydophila caviae | C. caviae ist typisch für Vertreter der Gattung Cavia (echte Meerschweinchen), zu denen auch das Hausmeerschweinchen gehört. Sie lässt sich bei ihnen in der Bindehaut des Auges nachweisen und verursacht eine Konjunktivitis.[4] |
| Chlamydophila pecorum | C. pecorum verursacht bei Koalas Erkrankungen des Urogenitaltrakts (Harn- und Geschlechtsorgane), die zur Unfruchtbarkeit führen können. Bei anderen Tierarten wird sie mit Fehlgeburten (Abort), Konjunktivitis, Enzephalomyelitis, Enteritis, Pneumonien und Polyarthritis in Verbindung gebracht.[4] |
| Chlamydia abortus (Chlamydophila abortus) | C. abortus verursacht bei Schafen und Ziegen den Chlamydienabort. Auch hier kann es sich um eine mit schweren Fieber einhergehende Zoonose handeln, in einzelnen Fällen gelang der Nachweis des Bakteriums bei Frauen, die berufsbedingt mit Schafen, speziell infizierten lammenden Muttschafen, zu tun haben und eine Fehlgeburt erlitten.[4][23] |
Schwangerschaft
| Klassifikation nach ICD-10 | |
|---|---|
| O98.3 | Sonstige Infektionen, hauptsächlich durch Geschlechtsverkehr übertragen, die Schwangerschaft, Geburt und Wochenbett komplizieren |
| A56.- | Sonstige durch Geschlechtsverkehr übertragene Chlamydienkrankheiten |
| P23.1 | Angeborene Pneumonie durch Chlamydien |
| P39.1 | Konjunktivitis durch Chlamydien beim Neugeborenen |
| ICD-10 online (WHO-Version 2019) | |
Chlamydien werden meist während des Geburtsvorganges von der Mutter auf das Kind übertragen. Eine Übertragung in der Gebärmutter ist möglich, aber selten. Eine Infektion kann bei dem neugeborenen Kind zu einer schweren Lungenentzündung und Augenentzündung, die eine Erblindung zur Folge haben kann, führen. Unbehandelt steigt das Risiko für eine Fehlgeburt, Totgeburt oder Frühgeburt.[24]
Therapie
Infektionen durch Chlamydien werden meist mit Tetracyclinen (z. B. Doxycyclin) behandelt oder mit Makroliden (z. B. Erythromycin oder Azithromycin) bei Schwangeren und Kindern. β-Lactam-Antibiotika wie Penicillin sind aufgrund der fehlenden Zellwand völlig unwirksam, da diese für ihren Wirkungsmechanismus eine Zellwand benötigen. Antibiotikaresistenzen sind bei Chlamydien eher selten, weshalb von der gut wirksamen Therapie mit Chinolon-Antibiotika, die teils mit unerwünschten Arzneimittelwirkungen behaftet ist, oft abgesehen wird. Weitere Informationen zu den Behandlungen sind bei den genannten Krankheiten aufgeführt. Es gibt keine Impfung gegen Chlamydien.[3] Derzeit laufen mehrere Studien.[25]
Literatur
- Karin D. Everett, Robin M. Bush, Arthur A. Andersen: Emended description of the order Chlamydiales, proposal of Parachlamydiaceae fam. nov. and Simkaniaceae fam. nov., each containing one monotypic genus, revised taxonomy of the family Chlamydiaceae, including a new genus and five new species, and standards for the identification of organisms. In: International journal of systematic bacteriology. Band 49, Nr. 2, April 1999, S. 415–440, ISSN 0020-7713. PMID 10319462.
- Michael T. Madigan, John M. Martinko, Jack Parker: Brock Mikrobiologie. Deutsche Übersetzung herausgegeben von Werner Goebel. 1. Auflage. Spektrum Akademischer Verlag GmbH, Heidelberg/Berlin 2000, ISBN 978-3-8274-0566-1.
- Helmut Hahn, Stefan H. E. Kaufmann, Thomas F. Schulz, Sebastian Suerbaum (Hrsg.): Medizinische Mikrobiologie und Infektiologie. 6. Auflage. Springer Verlag, Heidelberg 2009, ISBN 978-3-540-46359-7.
- Marianne Abele-Horn: Antimikrobielle Therapie. Entscheidungshilfen zur Behandlung und Prophylaxe von Infektionskrankheiten. Unter Mitarbeit von Werner Heinz, Hartwig Klinker, Johann Schurz und August Stich, 2., überarbeitete und erweiterte Auflage. Peter Wiehl, Marburg 2009, ISBN 978-3-927219-14-4, S. 189–191.
Weblinks
- Portal mit wissenschaftlichen Informationen zu Chlamydien (englisch). (Nicht mehr online verfügbar.) 9. Dezember 2011, archiviert vom Original am 27. Juni 2015; abgerufen am 26. November 2013.
- The comprehensive reference and education wiki on Chlamydia and the Chlamydiales. Chlamydiae.com
Einzelnachweise
- ↑ a b c d e f g Michael T. Madigan, John M. Martinko, Jack Parker: Brock Mikrobiologie. Deutsche Übersetzung herausgegeben von Werner Goebel, 1. Auflage. Spektrum Akademischer Verlag GmbH, Heidelberg/Berlin 2000, ISBN 978-3-8274-0566-1, S. 588–591.
- ↑ a b c d e f g h i Herbert Hof, Rüdiger Dörries: Duale Reihe: Medizinische Mikrobiologie. 3. Auflage. Thieme Verlag, Stuttgart 2005, ISBN 978-3-13-125313-2, S. 447–451.
- ↑ a b c d e f g h Helmut Hahn, Stefan H. E. Kaufmann, Thomas F. Schulz, Sebastian Suerbaum (Hrsg.): Medizinische Mikrobiologie und Infektiologie. 6. Auflage. Springer Verlag, Heidelberg 2009, ISBN 978-3-540-46359-7, S. 416–426.
- ↑ a b c d e f g h i j k l m n o p q r s K. D. Everett, R. M. Bush, A. A. Andersen: Emended description of the order Chlamydiales, proposal of Parachlamydiaceae fam. nov. and Simkaniaceae fam. nov., each containing one monotypic genus, revised taxonomy of the family Chlamydiaceae, including a new genus and five new species, and standards for the identification of organisms. In: International journal of systematic bacteriology. Band 49, Nr. 2, April 1999, S. 415–440, ISSN 0020-7713. PMID 10319462.
- ↑ David M. Ojcius, Toni Darville, Patrik M. Bavoil: Die heimliche Seuche. In: Spektrum der Wissenschaft. Nr. 2, Februar 2006. Spektrum der Wissenschaft Verlag, S. 28–35, ISSN 0170-2971.
- ↑ a b H. Okuda, K. Ohya u. a.: Detection of Chlamydophila psittaci by using SYBR green real-time PCR. In: The Journal of veterinary medical science / the Japanese Society of Veterinary Science. Band 73, Nr. 2, Februar 2011, S. 249–254, ISSN 0916-7250. PMID 20948172.
- ↑ TRBA (Technische Regeln für Biologische Arbeitsstoffe) 466: Einstufung von Prokaryonten (Bacteria und Archaea) in Risikogruppen. In: Website der Bundesanstalt für Arbeitsschutz und Arbeitsmedizin (BAuA). 25. April 2012, S. 54, abgerufen am 9. März 2013.
- ↑ a b c d e f g Chlamydiosen (Teil 2): Erkrankungen durch Chlamydophila psittaci, Chlamydophila pneumoniae und Simkania negevensis – RKI-Ratgeber. (Nicht mehr online verfügbar.) In: Webseite des Robert Koch-Instituts (RKI). 8. März 2010, archiviert vom Original am 16. Juli 2015; abgerufen am 31. Mai 2018. Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis.
- ↑ a b c d e Chlamydiosen (Teil 1): Erkrankungen durch Chlamydia trachomatis – RKI-Ratgeber. In: Webseite des Robert Koch-Instituts (RKI). 21. Dezember 2010, abgerufen am 31. Mai 2018.
- ↑ Prevalence and incidence of selected sexually transmitted infections. In: Webseite der Weltgesundheitsorganisation (WHO). WHO, Department of Reproductive Health and Research, 2011, abgerufen am 23. November 2013.
- ↑ a b S. Van Lent, J. R. Piet u. a.: Full genome sequences of all nine Chlamydia psittaci genotype reference strains. In: Journal of bacteriology. Band 194, Nr. 24, Dezember 2012, S. 6930–6931, ISSN 1098-5530. doi:10.1128/JB.01828-12. PMID 23209198. PMC 3510619 (freier Volltext).
- ↑ a b c d Jean Euzéby, Aidan C. Parte: Family Chlamydiaceae. In: List of Prokaryotic names with Standing in Nomenclature (LPSN). Abgerufen am 31. Mai 2018.
- ↑ R. S. Gupta, V. Bhandari, H. S. Naushad: Molecular Signatures for the PVC Clade (Planctomycetes, Verrucomicrobia, Chlamydiae, and Lentisphaerae) of Bacteria Provide Insights into Their Evolutionary Relationships. In: Frontiers in microbiology. Band 3, 2012, S. 327, ISSN 1664-302X. doi:10.3389/fmicb.2012.00327. PMID 23060863. PMC 3444138 (freier Volltext).
- ↑ a b J. Schachter, R. S. Stephens u. a.: Radical changes to chlamydial taxonomy are not necessary just yet. In: International journal of systematic and evolutionary microbiology. Band 51, Nr. 1, Januar 2001, S. 249; author reply S. 251–253, ISSN 1466-5026. doi:10.1099/00207713-51-1-249, PMID 11211265.
- ↑ R. S. Stephens, G. Myers u. a.: Divergence without difference: phylogenetics and taxonomy of Chlamydia resolved. In: FEMS immunology and medical microbiology. Band 55, Nr. 2, März 2009, S. 115–119, ISSN 1574-695X. doi:10.1111/j.1574-695X.2008.00516.x. PMID 19281563. (Review).
- ↑ a b Konrad Sachse, Patrik M. Bavoil u. a.: Emendation of the family Chlamydiaceae: Proposal of a single genus, Chlamydia, to include all currently recognized species. In: Systematic and Applied Microbiology. Band 38, Nr. 2, März 2015, S. 99–103, doi:10.1016/j.syapm.2014.12.004.
- ↑ G. Greub: International Committee on Systematics of Prokaryotes. Subcommittee on the taxonomy of the Chlamydiae: Minutes of the inaugural closed meeting, 21 March 2009, Little Rock, AR, USA. In: International journal of systematic and evolutionary microbiology. Band 60, Nr. 11, November 2010, S. 2691–2693, ISSN 1466-5034. doi:10.1099/ijs.0.028225-0. PMID 21048221.
- ↑ G. Greub: International Committee on Systematics of Prokaryotes. Subcommittee on the taxonomy of the Chlamydiae: Minutes of the closed meeting, 21 June 2010, Hof bei Salzburg, Austria. In: International journal of systematic and evolutionary microbiology. Band 60, Nr. 11, November 2010, S. 2694, ISSN 1466-5034. doi:10.1099/ijs.0.028233-0. PMID 21048222.
- ↑ G. Greub: International Committee on Systematics of Prokaryotes. Subcommittee on the taxonomy of Chlamydiae: Minutes of the closed meeting, 23 February 2011, Ascona, Switzerland. In: International journal of systematic and evolutionary microbiology. Band 63, Nr. 5, Mai 2013, S. 1934–1935, ISSN 1466-5026. doi:10.1099/ijs.0.028225-0.
- ↑ Matthias Horn, Michael Wagner: Bakterien, die in Acanthamöben leben: Dasein im Verborgenen. In: Biologie in unserer Zeit. Band 31, Nr. 3, Mai 2001, S. 160–168, doi:10.1002/1521-415X(200105)31:3<160::AID-BIUZ160>3.0.CO;2-T.
- ↑ Junge Menschen besonders von Chlamydien betroffen – BZgA informiert mit Social-Media-Kampagne. BZgA, 4. Oktober 2021, abgerufen am 14. September 2022.
- ↑ Factsheet für Ärztinnen und Ärzte zur Chlamydien-Infektion, Bundeszentrale für gesundheitliche Aufklärung (BZgA), 2021.
- ↑ Marianne Abele-Horn (2009), S. 191.
- ↑ Chlamydien in der Schwangerschaft. In: Webseite HealthExpress. 31. Januar 2017, abgerufen am 26. Februar 2017.
- ↑ Zwei Impfstoffe gegen Chlamydien bei Mäusen erfolgreich getestet. In: Deutsches Ärzteblatt. Deutscher Ärzteverlag GmbH, Redaktion Deutsches Ärzteblatt, 26. Juli 2016, abgerufen am 31. Mai 2018.
