Einzelmolekülexperiment
Ein Einzelmolekülexperiment (englisch: Single-molecule experiment) ist ein Experiment, das die Eigenschaften von Einzelmolekülen betrachtet. Einzelmolekül-Experimente stehen im Gegensatz zu Messungen einer Gesamtheit oder Ansammlung von Molekülen, bei denen man das Verhalten einzelner Moleküle nicht unterscheiden kann und nur Durchschnittswerte gemessen werden. Während viele Experimente in der Biologie, Chemie oder Physik nicht empfindlich genug sind, Einzelmoleküle zu beobachten, gibt es Einzelmolekül-Fluoreszenztechniken, die große Begeisterung auslösten, da sie neue Details erkennen lassen, die mit den früheren Messtechniken nicht sichtbar waren. In der Tat sind seit den 1990er Jahren viele Techniken entwickelt worden, Einzelmoleküle zu detektieren.[1][2]
Die ersten Einzelmolekülexperimente waren patch-clamp-Experimente, die in den 70er Jahren durchgeführt wurden, aber diese beschränken sich auf Messungen an Ionenkanälen. Heute verwendet man Einzelmolekül-techniken, die die Bewegung von Myosin auf Aktinfilamenten im Muskelgewebe messen können und untersucht die spektroskopischen Details der Umgebung in Lösungen. Biologische Polymere wurden mit Rasterkraftmikroskopen untersucht. Mikroskopisch werden, normalerweise im Polymer, einzelne Moleküle und deren elastische Verformung gemessen.
Geschichte
Einzelmolekülexperimente wurden schon über Jahrzehnte bei niedrigem Druck durchgeführt, aber erst seit 1989 von William E. Moerner und Lothar Kador auch in kondensierten Phasen.[3] Ein Jahr später waren Michel Orrit und Jacky Bernard in der Lage, die Absorption von Einzelmolekülen anhand ihrer Fluoreszenz zu detektieren.[4] Es gibt viele Techniken, Einzelmoleküle zu beobachten, wie beispielsweise die Massenspektrometrie, wo einzelne Ionen detektiert werden. Zusätzlich gibt es Einzelmoleküldetektion auf dem Gebiet der Ionenkanäle mit der Entwicklung der patch clamp Technik von Erwin Neher und Bert Sakmann. Die Idee, mit Hilfe von Leitfähigkeitsmessungen Einzelmoleküle nachzuweisen, stieß an ihre Grenzen. Fluoreszenzmessungen dagegen sind geeignet, um ein Einzelmolekül zu beobachten, dank der Sensibilität von optischen Detektoren, die in der Lage sind, einzelne Photonen zu zählen. Die Beobachtung einzelner Moleküle mit spektroskopischen Methoden erfordert eine isolierte Umgebung und die Emission von Photonen bei photochemischer Anregung, wozu eine Ausstattung (Photomultiplier oder erweiterte Photodioden) gehört, mit der man in der Lage ist, Photoemissionen mit hoher Sensibilität und hoher Zeitauflösung aufzunehmen.
Kürzlich war Einzelmolekülfluoreszenz Gegenstand des Interesses für biologische Bildgebung, da das Markieren von Biomolekülen wie Proteinen und Nucleotiden zum Erforschen der enzymatischen Funktionen nicht in einer Gesamtstruktur erfolgen kann wegen der feinen zeitabhängigen Bewegungen bei der Katalyse und strukturellen Reorganisation. Das am häufigsten untersuchte Protein ist das Myosin/Actin-Enzym, das man in Muskelfasern findet. Durch die Einzelmolekültechnik wurden viele einzelne Reaktionsschritte beobachtet und bei vielen dieser Proteine charakterisiert.
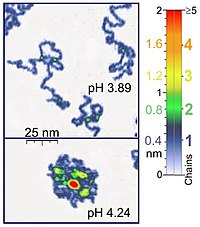
Rasterkraftmikroskope (atomic force microscope (AFM)) sind auch für Einzelmolekülexperimente von biologische Bedeutung, weil sie in der gleichen Größenordnung arbeiten, wie die meisten biologischen Polymere. Außerdem lassen sich AFMs für synthetische Polymere verwenden, und sie können Polymerketten in 3D abbilden. das AFM ist fein genug, um absorbierte Polyelektrolyte (bspw. Poly-2-vinylpyridin)in Lösung zu beobachten. Überlagern sich zwei Ketten, so hat man die doppelte Dicke einer Einzelkette. Bei richtiger Einstellung der Scan-Parameter bleibt die Konformation der Ketten für Stunden erhalten und erlaubt die Durchführung der Experimente in Lösung unter den geeigneten Reaktionsbedingungen.[1] Kontrolliert man das Kraftfeld um die Probe herum, so erhält man hochaufgelöste Bilder.[5][6] Optische Pinzetten sind auch verwendet worden, um DNA-Protein-Wechselwirkungen zu messen.[5][6]
Experimente
Konzept
Einzelmolekülfluoreszenzspektroskopie nutzt die Fluoreszenz eines Moleküls, um Informationen über die Umgebung, Struktur und Position zu gewinnen. Die Technik eröffnet die Möglichkeit, Informationen zu gewinnen, die man nicht durch Mittelwertbildung einer Gesamtstruktur erhält. In den meisten Experimenten mit einzelnen Molekülen sind die Ergebnisse gemittelt.
Einzel-Kanal-Aufnahme
Wie im Fall der Einzelmolekülfluoreszenzspektroskopie wird diese Technik, bekannt als Einzelkanalaufnahme, angewandt, um spezifische kinetische Informationen – der Ionenkanalfunktion – die nicht aus der Gesamtstruktur ablesbar sind, bspw. Zellaufnahmen, aufzunehmen.[7] Ionenkanäle wechseln zwischen leitenden und nichtleitenden Zuständen, je nach Konformation. Daher kann der funktionelle Zustand eines Ionenkanals gemessen werden. Man führt Einzelmolekülexperimente systematisch unter verschiedenen Reaktionsbedingungen durch, so dass man die verschiedenen kinetischen Zustände des Ionenenkanals betrachten kann.[7]
Biomolekülmarkierung
Einzelne Fluorophore können chemisch an Biomoleküle angeknüpft werden, wie beispielsweise Proteine und DNA, und die Bewegung der individuellen Moleküle kann mit Hilfe des Fluoreszenzmarkers aufgezeichnet werden. Molekülbewegungen können detektiert werden anhand der Änderung der Emissionsintensität oder Strahlungsdauer, die auf Änderungen in der Umgebung hinweist. Beispielsweise hat Einzelmolekülmarkierung viel Informationen geliefert über Kinesinproteine in Mikrotubuli von Muskelzellen.
Einzelmolekül-Fluoreszenzresonanz-Energietransfer (FRET)
Im Einzelmolekül-Fluoreszenz-Resonanz-Energietransfer (FRET) wird das Molekül an zwei Stellen markiert. Ein Laserstrahl wird zur Anregung auf die erste Probe eingestrahlt. Sobald diese Probe relaxiert und ein Photon emittiert, kann es die andere Probe photochemisch anregen. Die Effizienz der Absorption des emittierten Photons der ersten Probe durch die zweite Probe hängt von der Entfernung zwischen den Proben ab. Ändert sich die Entfernung in Abhängigkeit von der Zeit, dann zeigt das Experiment die interne Dynamik des Moleküls.
Einzelmolekül-Experiment gegenüber Gesamtexperiment
Sieht man sich die Daten individueller Moleküle an, so kann man kinetische Funktionen aufstellen (1. und 2. Ordnung), wobei man aus der Gesamtfunktion den Zerfall der zugeordneten Einzelfunktion erhält. Aus einer einzelnen Funktion kann man Rückschlüsse ziehen, wie sich ein System verhält. (s. Reaktionskinetik)[8][9] Im Besonderen kann man den Reaktionsweg eines Enzyms aufzeichnen, wenn man die Aktivität eines Einzelenzyms aufnimmt.[10] Zusätzlich sind verschiedene Methoden erwähnt, wie man Einzelmoleküle analysiert. Darüber hinaus werden von verschiedenen Autoren unterschiedliche Aspekte der Datenauswertung genannt (lineare Anpassungen, Statistik) und es gibt genügend Ziele bei der Analyse von Einzelmolekülen, wie beispielsweise eine Umgebung ohne „Rauschen“ bei der Aufnahme, Filtern der Lösung, genügend Zeit zur Datenanalyse.
Ziel
Einzelmoleküldetektion betrifft die Optik, Elektronik, Biologie und Chemie. In biologischen Systemen war die Beobachtung von Proteinen und anderen komplexen biologischen Strukturen begrenzt auf eine Gesamtstruktur und es war nahezu unmöglich, die Kinetik einzelner Moleküle zu beobachten. Erst nachdem die Einzelmolekülfluoreszenzmikroskopie angewandt wurde, um beispielsweise Kinesin-Myosin-Paare im Muskelgewebe zu beobachten, wurde der Bewegungsapparat verstanden. Diese Experimente waren jedoch meistens auf Reagenzglasversuche beschränkt bis nützliche Techniken für die Messungen in lebenden Zellen geschaffen wurden. Die Aussicht auf in vivo Einzelmoleküldetektion[11] verbirgt das enorme Potential, Biomoleküle in lebenden Organismen zu beobachten. Diese Techniken werden oft angewendet, um Proteine zu erforschen. Die Technik lässt sich auf andere Gebiete der Chemie ausweiten, beispielsweise heterogene Oberflächen.[12]
Einzelnachweise
- ↑ a b c Y. Roiter and S. Minko: AFM Single Molecule Experiments at the Solid-Liquid Interface: In Situ Conformation of Adsorbed Flexible Polyelectrolyte Chains. In: Journal of the American Chemical Society, 127, S. 15688–15689 (2005). doi:10.1021/ja0558239
- ↑ MF Juette, DS Terry, MR Wasserman, Z Zhou, RB Altman, Q Zheng, SC Blanchard: The bright future of single-molecule fluorescence imaging. In: Curr Opin Chem Biol. 20, Juni 2014, S. 103–11. doi:10.1016/j.cbpa.2014.05.010. PMID 24956235. PMC 4123530 (freier Volltext).
- ↑ W. E. Moerner and L. Kador: Optical detection and spectroscopy of single molecules in a solid. In: Phys. Rev. Lett. 62, 2535–2538 (1989). doi:10.1103/PhysRevLett.62.2535
- ↑ M. Orrit and J. Bernard: Single pentacene molecules detected by fluorescence excitation in a p-terphenyl crystal. In: Phys. Rev. Lett. 65, 2716–2719 (1990). doi:10.1103/PhysRevLett.65.2716
- ↑ a b D. Murugesapillai et al.: DNA bridging and looping by HMO1 provides a mechanism for stabilizing nucleosome-free chromatin. In: Nucl Acids Res (2014) 42 (14): 8996-9004, doi:10.1093/nar/gku635
- ↑ a b D. Murugesapillai et al.: Single-molecule studies of high-mobility group B architectural DNA bending proteins. In: Biophys Rev. 2016. doi:10.1007/s12551-016-0236-4.
- ↑ a b B. Sakmann and E. Neher, Single-Channel Recording, ISBN 978-0-306-41419-0 (1995).
- ↑ O. Flomenbom, and R. J. Silbey: Utilizing the information content in two-state trajectories (Memento des Originals vom 14. Januar 2012 im Internet Archive) Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis., Proc. Natl. Acad. Sci. USA 103, 10907–10910 (2006).
- ↑ O. Flomenbom, and R. J. Silbey: Toolbox for analyzing finite two-state trajectories. Phys. Rev. E 78, 066105 (2008); arxiv:0802.1520.
- ↑ O. Flomenbom, K. Velonia, D. Loos, et al.: Stretched exponential decay and correlations in the catalytic activity of fluctuating single lipase molecules (Memento des Originals vom 14. Januar 2012 im Internet Archive) Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis., Proc. Natl. Acad. Sci. US 102, 2368–2372 (2005).
- ↑ Hong Zhan, Ramunas Stanciauskas, Christian Stigloher, Kevin K. Dizon, Maelle Jospin, Jean-Louis Bessereau, Fabien Pinaud: In vivo single-molecule imaging identifies altered dynamics of calcium channels in dystrophin-mutant C. elegans. In: Nature Communications. 5, 2014, S. ncomms5974. bibcode:2014NatCo...5E4974Z. doi:10.1038/ncomms5974.
- ↑ R. Walder, N. Nelson, D. K. Schwartz: Super-resolution surface mapping using the trajectories of molecular probes. In: Nature Communications. 2, 2011, S. 515. bibcode:2011NatCo...2E.515W. doi:10.1038/ncomms1530.