De-Novo-Peptidsequenzierung
Die De-Novo-Peptidsequenzierung ist ein biochemisches Verfahren zur Bestimmung der Aminosäuresequenz von Proteinen oder Peptiden per Tandem-Massenspektrometrie (MS/MS).[1] Sie ist eine Methode zur Proteinsequenzierung.
Eigenschaften
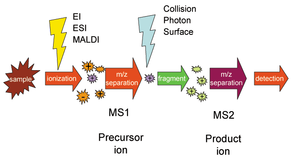
Im Vergleich zu den üblichen massenspektrometrischen Verfahren zur Proteincharakterisierung erfolgt bei der De-Novo-Peptidsequenzierung eine zweite Fragmentierung der Peptide.[2] Diese zusätzliche Fragmentierung kann in einem Reflektron z. B. durch einen kollisionsinduzierten (CID) oder durch einen LASER-induzierten Post-Source Decay (PSD) erreicht werden. Bei mehrfach geladenen Fragmenten kann die weitere Fragmentierung auch durch eine Electron Capture Dissociation oder durch eine Elektronenstoßionisation erreicht werden.[3][1] Im Spektrum erhält man aus jedem Fragment der ersten Runde verschiedene Fragmente weniger Aminosäuren, die durch Aneinanderreihung anhand der Überlappungen die Peptidsequenz ergeben. Dadurch kann der Aufbau der Peptidfragmente im Sinne einer N-terminalen Peptidsequenzierung ermittelt werden.[4] Da diese massenspektrometrische Methode nicht auf Informationen über die Aminosäuresequenz angewiesen ist,[5] wird sie als de novo bezeichnet.
Einzelnachweise
- ↑ a b K. F. Medzihradszky: Peptide sequence analysis. In: Methods in enzymology. Band 402, 2005, Skriptfehler: Das Modul gab einen nil-Wert zurück. Es wird angenommen, dass eine Tabelle zum Export zurückgegeben wird., S. 209–244, Modul:Vorlage:Handle * library URIutil invalid, PMID 16401511.
- ↑ J. Seidler, N. Zinn, M. E. Boehm, W. D. Lehmann: De novo sequencing of peptides by MS/MS. In: Proteomics. Band 10, Nummer 4, Februar 2010, Skriptfehler: Das Modul gab einen nil-Wert zurück. Es wird angenommen, dass eine Tabelle zum Export zurückgegeben wird., S. 634–649, Modul:Vorlage:Handle * library URIutil invalid, PMID 19953542.
- ↑ J. P. Reilly: Ultraviolet photofragmentation of biomolecular ions. In: Mass spectrometry reviews. Band 28, Nummer 3, 2009, Skriptfehler: Das Modul gab einen nil-Wert zurück. Es wird angenommen, dass eine Tabelle zum Export zurückgegeben wird., S. 425–447, Modul:Vorlage:Handle * library URIutil invalid, PMID 19241462.
- ↑ J. W. Morgan, J. M. Hettick, D. H. Russell: Peptide sequencing by MALDI 193-nm photodissociation TOF MS. In: Methods in enzymology. Band 402, 2005, Skriptfehler: Das Modul gab einen nil-Wert zurück. Es wird angenommen, dass eine Tabelle zum Export zurückgegeben wird., S. 186–209, Modul:Vorlage:Handle * library URIutil invalid, PMID 16401510.
- ↑ J. Allmer: Algorithms for the de novo sequencing of peptides from tandem mass spectra. In: Expert review of proteomics. Band 8, Nummer 5, 2011, Skriptfehler: Das Modul gab einen nil-Wert zurück. Es wird angenommen, dass eine Tabelle zum Export zurückgegeben wird., S. 645–657, Modul:Vorlage:Handle * library URIutil invalid, PMID 21999834.