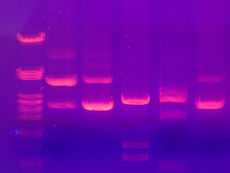
Gelelektrophorese


Gelelektrophorese (Wortteile: Gel|elektro|phorese – letzterer abgeleitet von altgriechisch φέρειν pherein ‚tragen‘) ist eine analytische Methode der Chemie und Molekularbiologie, um verschiedene Arten von Molekülen zu trennen.
Prinzip
Die unterschiedliche Ionenbeweglichkeit wird in verschiedenen Elektrophorese-Methoden genutzt, um ionische Substanzen im elektrischen Feld zu trennen und z. B. getrennt einer Messung zuzuführen.
Bei der Gelelektrophorese wandert eine Mischung aus zu trennenden, elektrisch geladenen Molekülen unter Einfluss eines elektrischen Felds durch ein Gel, welches in einer ionischen Pufferlösung (Elektrophoresepuffer) liegt. Je nach Größe und Ladung der Moleküle bewegen sich diese unterschiedlich schnell durch das als Molekularsieb wirkende Gel. Dabei wandern kleine, negativ geladene Moleküle (Anionen) am schnellsten in Richtung der positiv geladenen Anode und positiv geladene Moleküle (Kationen) in Richtung der negativ geladenen Kathode. Die zugrunde liegenden Theorien sind die sich ergänzenden Ogston Siebtheorie und die Reptationstheorie.[1][2][3] Während die Siebtheorie das Zurückhalten (synonym Retention) von sphärischen Makromolekülen (z. B. Proteine oder Micellen) durch eine definierte Porosität der Gelmatrix beschreibt, handelt die Reptationstheorie von einer Retention von Makromolekülen durch Reibung nichtsphärischer Makromoleküle an der Gelmatrix (z. B. DNA und RNA).
Gel-Matrix
Die Polymermoleküle des Gels bilden ein mehr oder weniger engmaschiges, dreidimensionales Gitter, das die Migration (Wanderung) der zu trennenden Moleküle im elektrischen Feld mehr oder weniger verlangsamt.
Agarose
Agarosegele sind relativ großporig (150 nm bei einprozentigen, 500 nm bei 0,16-prozentigen Gelen) und eignen sich gut zur Trennung von DNA und hochmolekularen Proteinen. Die Distanz zwischen DNA-Banden unterschiedlicher Länge ist abhängig von der Konzentration an Agarose im Gel. Höhere Konzentrationen erfordern längere Laufzeiten (manchmal sogar Tage).[4] Haupteinsatzgebiet ist jedoch die Trennung von Nukleinsäuren. Agarosegele werden aus den natürlichen Polysacchardipolymeren aus Seetang hergestellt. Bei der Elektrophorese mit Agarosegel handelt es sich um ein physikalisches Setting. Nach dem Experiment kann das Ergebnis mithilfe eines Plastikbeutels tiefgekühlt gelagert werden.[5]
Polyacrylamid
Gele aus Polyacrylamid werden durch Polymerisation von Acrylamid hergestellt. Sie weisen wesentlich kleinere Poren auf (3–6 nm). Die Porengröße hängt von der Acrylamidkonzentration und dem Vernetzungsgrad ab. Häufig werden hiermit Proteine zwischen 5 und 20.000 kDA getrennt.
Stärke
Eine weitere Möglichkeit bildet die Verwendung von teilweise hydrolysierter Kartoffelstärke in Konzentrationen zwischen 5 % und 10 %. Es handelt sich dabei um ein untoxisches Medium für die Elektrophorese von nicht-denaturierten Proteinen. Die Auftrennung erfolgt nach Größe und Ladung und die Visualisierung erfolgt durch Naphthol-Schwarz- oder Amido-Schwarz-Färbung.[6][7] Ohne Zugabe von Bioziden neigen Stärkegele zur mikrobiellen Zersetzung.
Durchführung
Die klassische Gelelektrophorese wird als Zonenelektrophorese durchgeführt. Eine Methode zur Erzielung einer höheren Auflösung ist die diskontinuierliche Elektrophorese.
Bei der Gelelektrophorese entsteht Wärme. Diese muss abgeführt werden, um optimale Bedingungen zu gewährleisten. Deswegen sollte die Gelelektrophorese in gekühlten Apparaturen bei konstanten Temperaturen durchgeführt werden, um reproduzierbare Ergebnisse zu erzielen.
Im Idealfall wird die Elektrophorese beendet, wenn die kleinsten beziehungsweise mobilsten Moleküle das Ende des Gels erreicht haben. Das garantiert die höchstmögliche Auftrennung der Moleküle.
Auswertung
Gleiche Moleküle laufen in diskreten Zonen – umgangssprachlich als Banden bezeichnet – durch das Gel. Mehrere Proben können parallel nebeneinander gleichzeitig durch dasselbe Gel laufen. Ist die Größe einiger Moleküle bekannt, kann man durch Vergleich von deren Banden mit den restlichen Banden die Größe der anderen Moleküle abschätzen. Solche Molekülmassenstandards sind kommerziell erhältlich. Ähnlich funktioniert auch ein Komigrationsstandard, mit dessen Hilfe eine unbekannte mit einer bekannten Probenzusammensetzung verglichen wird. Als Molekülmassenstandards werden DNA oder Proteine verwendet.
Eine Bestimmung der Menge einer Substanz in einer Bande beziehungsweise der relative Anteil einer Bande (siehe: Quantifizierung) ist nach der Färbung und Fotografie oder Scan des Gels und einer anschließenden densitometrischen Auswertung möglich, mit der Einschränkung, dass bei sehr dunklen Banden der innere Bereich der Bande mangels Lichteinstrahlung nicht mit gewertet werden kann. Zur Bestimmung der Messwerte eines Geles wie z. B. Laufweiten, Molekülmassen, Quantifizierungen oder Normalisierung wird in den meisten Fällen eine Auswertungssoftware genutzt.
Zur Auswertung des Gels nach der Elektrophorese werden die zu trennenden Moleküle entweder vor der Elektrophorese radioaktiv markiert und anschließend in einer Autoradiographie nachgewiesen oder nach der Elektrophorese mit verschiedenen Farbstoffen versetzt.
Bei der Nukleotidanalytik wird häufig Ethidiumbromid verwendet, das mit Nukleinsäuren interkaliert und diese unter UV-Licht sichtbar macht. Proteine lassen sich mit Proteinfarbstoffen direkt anfärben, z. B. mit Coomassie-Brillant-Blau oder im Zuge der Silberfärbung. Eine Alternative zur Färbung ist das anschließende Blotting. Man unterscheidet:
- Western Blot (immunologischer Nachweis von Proteinen mit markierten Antikörpern)
- Southern Blot (Nachweis von DNA durch Hybridisierung mit DNA- oder RNA-Sonden)
- Northern Blot (Nachweis von mRNA ebenfalls durch Hybridisierung mit Nukleotidsonden)
Einsatzgebiete
Gelelektrophoresen finden in der Molekularbiologie, Biochemie und Lebensmittelanalytik Anwendung. Gele können ohne großen Aufwand selbst hergestellt werden. Fertige Gele und die entsprechenden Puffersysteme können zudem kommerziell erworben werden.
Es existieren zahlreiche Spezialanwendungen:
- SDS-PAGE zur Trennung von Stoffgemischen (häufig Proteinen) nach Molekülgröße
- IEF zur Trennung von Proteinen nach ihrem isoelektrischem Punkt
- 2D-Gelelektrophorese als Kombination aus SDS-Page und IEF für komplexe Proteingemische
- Diskontinuierliche Elektrophorese
- QPNC-PAGE zur Analytik von Metalloproteinen
- Nativ-Gelelektrophorese zur Untersuchung der Proteinfaltung
- SDD-AGE zur Untersuchung von Proteinaggregaten
- Pulsed-Field-Gelelektrophorese (PFGE) zur Trennung großer DNA-Fragmente
- Kapillarelektrophorese (CE)
Literatur
- Friedrich Lottspeich, Haralabos Zorbas: Bioanalytik. Spektrum Akademischer Verlag, Heidelberg u. a. 1998, ISBN 3-8274-0041-4.
- Hubert Rehm, Thomas Letzel: Der Experimentator: Proteinbiochemie / Proteomics. 6. Auflage. Spektrum Akademischer Verlag, Heidelberg 2010, ISBN 978-3-8274-2312-2.
- D. E. Garfin: One-dimensional gel electrophoresis. In: Methods in enzymology. Band 182, 1990, S. 425–441, PMID 2314252.
- D. E. Garfin: One-dimensional gel electrophoresis. In: Methods in enzymology. Band 463, 2009, S. 497–513, doi:10.1016/S0076-6879(09)63029-9, PMID 19892189.
Weblinks
Einzelnachweise
- ↑ Alexander George Ogston: The spaces in a uniform random suspension of fibres. In: Transactions of the Faraday Society. Bd. 54, 1958, ISSN 0014-7672, S. 1754–1757, doi:10.1039/TF9585401754.
- ↑ Gary W. Slater, Jean Rousseau, Jaan Noolandi, Chantal Turme, Marc Lalande: Quantitative analysis of the three regimes of DNA electrophoresis in agarose gels. In: Biopolymers. Bd. 27, Nr. 3, 1988, ISSN 0006-3525, S. 509–524, PMID 3359012, doi:10.1002/bip.360270311.
- ↑ Oscar J. Lumpkin, Philippe Déjardin, Bruno H. Zimm: Theory of gel electrophoresis of DNA. In: Biopolymers. Bd. 24, Nr. 8, 1985, S. 1573–1593, PMID 4041551, doi:10.1002/bip.360240812.
- ↑ Agarose Gel Electrophoresis. Abgerufen am 18. Februar 2015
- ↑ Joseph Sambrook, David Russell: Molecular Cloning - A Laboratory Manual
- ↑ Gordon A.H.: Electrophoresis of proteins in polyacrylamide and starch gels. American Elsevier Publishing Company, Inc, New York 1975.
- ↑ Smithies O.: Zone electrophoresis in starch gels: group variations in the serum proteins of normal adults. In: Biochem. J. Band 61, Nr. 4, 1955, S. 629–641, PMID 13276348, PMC 1215845 (freier Volltext).