Benutzer:Jonas Honold/Artikelentwurf
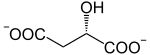
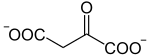
Das Enzym Malatdehydrogenase (MDH) katalysiert die reversible Oxidation von L-Malat zu Oxalacetat. Sie ist im Citratzyklus, im Aspartatzyklus und beim Energietransport durch die Malat-Aspartat-Shuttle beteiligt und damit unverzichtbar im Stoffwechsel aller aerober Lebewesen. Im Gegensatz zu Prokaryoten, welche keine Isozyme der Malatdehydrogenase enthalten, existieren in Eukaryoten mitochondriale (MDHM) und zytosolische (MDHC) Isoformen. Pilze und Pflanzen besitzen eine in den Glyoxisomen lokalisierte Malatdehydrogenase, die am Glyoxylatzyklus beteiligt ist. Darüber hinaus sind in den Chloroplasten pflanzlicher Zellen noch eine Form des Enzyms, die essentielle Aufgaben im Calvin-Zyklus und dem spezialisierteren C4-Zyklus bzw. Crassulaceen-Säurestoffwechsel übernimmt.
Konformationelle Gemeinsamkeiten und Ähnlichkeiten in der Struktur der umzusetzenden Substrate legen eine enge Verwandtschaft zwischen MDG und LDH nahe.
Während Prokaryoten eine einzige Form des Enzyms besitzen, haben alle Eukaryoten zwei Isozyme, die in den Mitochondrien und im Zytoplasma lokalisiert sind (MDHM und MDHC). Pilze und Pflanzen besitzen außerdem noch eine Form in den Glyoxisomen, die am Glyoxylatzyklus beteiligt ist. Zuletzt haben Pflanzen in ihren Chloroplasten noch eine Form des Enzyms, die essentielle Aufgaben im Calvin-Zyklus und dem spezialisierteren C4-Zyklus bzw. Crassulaceen-Säurestoffwechsel übernimmt. Die MDH von Archaeen dagegen ist eng verwandt mit der Lactatdehydrogenase.
Das mitochondriale Enzym wird allosterisch durch Citrat reguliert.
Alle Isoformen der MDH benötigen NAD als Coenzym, bis auf pflanzliche MDH, die NADP als Coenzym[1]
Katalysierte Reaktion
Die katalytisch Umsetzung von Malat ähnelt der des L-Lactats mittels Laktatdehydrogenase. Das Coenzym NAD+ fungiert als Oxidationsmittel.
Isoformen
Es existieren zahlreiche Isoformen der Malatdehydrogenase, darunter:
| Name/Abk. | Lokalisierung | Übergeordnetes Taxon | ENZYM-Eintrag | UniProt-Eintrag | Länge (Aminosäuren) |
|---|---|---|---|---|---|
| MDH/MDHC | Zytoplasma | Lebewesen | EC 1.1.1.37 | UniProt P40925 (Mensch) | 333 |
| MDHM | Mitochondrien | Eukaryoten | EC 1.1.1.37 | UniProt P40926 (Mensch) | 338 |
| MDHG | Glyoxysomen | Pflanzen | EC 1.1.1.37 | UniProt Q9ZP05 (Arabidopsis) | 354 |
| MDHP | Chloroplasten | Pflanzen | EC 1.1.1.82 | UniProt Q9SN86 (Arabidopsis) | 403 |
| MDH | Zytoplasma | Archaeen | EC 1.1.1.37 | UniProt Q9V0D5 (Pyrococcus) | 362 |
MsMDH aus Metallosphaera sedula kann sowohl NAD+ als auch NADP+ als Cofaktor nutzen. (https://www.sciencedirect.com/science/article/abs/pii/S0006291X19300245)
Isoformen der MDG weisen trotz unterschiedlicher Primärstruktur, welche teils nur eine 20 %ige Übereinstimmung aufweist, ähnliche Konformation auf.
mitochondriale Isoform ähnelt der prokaryotischen MDG
Einige prokaryotische Malatdehydrogenasen besitzen in ihrer Aminosäuresequenz große Übereinstimmungen mit den eukaryotischen Isoformen. So ähnelt die MDH aus E. coli der mitochondrialen MDHM, während das aus Thermus flavus stammende Enzym besonders große Ähnlichkeiten zur eukaryotischen MDH aus dem Cytosol und den Chloroplasten aufweist. Dies weist auf einen endosymbiotischen Ursprung eukaryotischer MDH hin.
MDH sind eng verwandt mit der Laktatdehydrogenase, weisen Ähnlichkeiten in Struktur und katalytischer Wirkung auf. Die größten Übereinstimmungen bestehen bei MDH aus Archaeen (z. B. Haloarcula marismortui), evolutionärer Link zwischen Laktatdehydrogenase und MDH
(https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2142602/pdf/7849603.pdf)
Eigenschaften
mitochondriale MDH unterscheidet sich in Substrat Affinität und pH-Optimum von zytosolischer MDH. Der Anteil an mitochondrialer und zytosolischer MDH variiert, je nach Organ. Im Myokard überwiegt der Anteil zytosolischer MDH, wohingegen
Die zytosolische Enzymaktivität ist in der Leber am größten, gefolgt von Herz Skelettmuskel und Gehirn.
Jede Untereinheit besitzt eine N-Terminale NAD-bindenden Domäne und eine Malat-bindende Domäne am C-terminalen Ende.
Untereinheiten sind autonom, weisen keine Kooperativität auf.
Drei Ariginin-Aminosäurereste an Stelle 102, 109 und 171 spielen in der Bindung des Substrats und deren katalytischer Umsetzung eine entscheidende Rolle und sind daher in allen MDHs enthalten.
Enzymregulation
MDH wird allosterisch durch Citrat reguliert.
Struktur
Homodimer oder Homotetramer, die Proteinuntereinheiten besitzen eine Masse von 30 bis 40 kDa.