Diskussion:Polymerase-Kettenreaktion/Archiv
Erster Artikel
In dieser Version. --91.5.216.127 15:43, 29. Mai 2008 (CEST)
- Er ist wohl nicht der erste Artikel. MfG Harry8 00:48, 18. Jun. 2012 (CEST)
- Siehe WP:ME! MfG Harry8 00:49, 18. Jun. 2012 (CEST)
Nested PCR
Warum gibts im gesamten Wikipedia, und selbst als Untereinheit im Artikel PCR, keine Angaben über die Nested-PCR und weitere PCR Methoden???? (nicht signierter Beitrag von 92.227.189.226 (Diskussion | Beiträge) 22:42, 18. Sep. 2009 (CEST))
Sonstiges
Hallo, ich bin zum ersten mal hier in wikipedia aktiv und weiss nicht, ob das der richtige weg ist, wenn einem etwas nicht ganz stimmig in einem artikel erscheint, schreibe es aber einfach trotzdem mal hier rein:
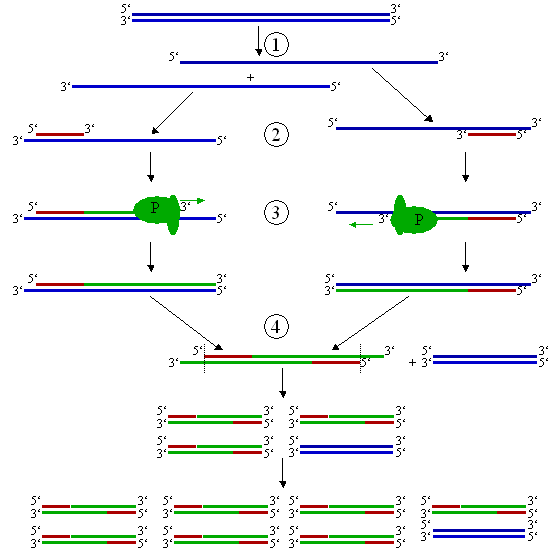
In der Abbildung zur Polymerase-Kettenreaktion ist ein Schritt 4 dargestellt, der den Eindruck vermittelt, dass sich am Ende des ersten Zyklus die ursprünglichen Matrizenstränge von den neu synthetiesierten (einseitig terminierten) Nukleinsäuresträngene trennen und sich jeweils die alten Matrizenstränge wieder zusammenlagern sowie die neu synthetisierten Stränge hybridisieren. Das ist falsch.
Im übrigen finde ich den Artikel prima ! Viele Grüße, Dagmar
Diskussionvorschlag: Das Bild von dem Eppendorf cycler sieht mir etwas nach Product-placement aus.. viele Grüße M (nicht signierter Beitrag von 2001:4ca0:4103:b:f2de:f1ff:fe20:92c3 (Diskussion) 18. Juni 2012)
Anwendungen in eigene Artikel
Mein Vorschlag wäre, die einzelnen Anwendungen auszulagern. Für die meisten gibt es sowieso schon eigene Artikel, wo die Methodik noch einmal beschrieben wird. Hier könnten sie deshalb nur kurz (in einem Satz) dargestellt und verlinkt werden. --Nina 13:04, 27. Jan 2005 (CET)
Gliederung
Die Abschnitte "Ablauf", "Beispiel" und "Schritte des PCR-Prozesses" sind teilweise redundant, zumindest aber sehr schlecht aufeinander abgestimmt und wenig zielführend. Der Abschnitt "PCR in der Praxis" wirkt verfehlt, ohne einen Abschnitt "PCR in der Theorie" macht der Titel auch nicht viel Sinn. Dahingegen fehlt bei Ablauf das wesentliche an der PCR, nämlich weshalb letztendlich mithilfe von zwei Primern eine ganz bestimmte Sequenz abgelesen werden kann, obwohl doch die Polymerase immer ganze Stränge abarbeitet und lediglich bei einem Primer anfängt. Die Abbildung zeigt das ja teilweise, nur der Text lässt diese Frage offen. --Saperaud ☺ 02:44, 28. Nov 2005 (CET)
nein ich finde die gliederung sehr gut, jedoch würde ich gerne wissen wieso die DNA-Polymerase bei 70°C sich anlagert. sobald doch die einzelstränge vorliegen und die primer daran gebunden sind kann doch die Polymerase anfangen zu arbeiten. ist es aufgrund dessen, dass die reaktion schlicht weg schneller von statten geht (RGT-Regel...).ich bitte um aufklärúng ;) danke schon mal. PS der artikel ist wirklich super + hoffentlich habe ich es nicht überlesen ;) mfg MauZi
Geschichte
Ist nicht schlüssig. (1.)Zuerst wird erzählt, dass Mullis orinär "Taq" verwendet hätte, (2.)dann, dass er noch "keine stabile Polymerase" hatte und das Verfahren ineffektiv gewesen sei, (3.)dann, daß man heute von thermophilen Bakterien das Enzym nimmt, dass deshalb "Taq" heissen würde. Was denn nun wirklich in welcher Reihenfolge? --Wikipit 11:48, 4. Jan 2006 (CET)
Kritik an der Definition
Ohne lebende Organismen, sicher irgendwie. Nur die Enzyme werden genau lebenden Organismen entnommen oder werden sie synthetisiert? Kann man das präziser fassen? --Wikipit 12:02, 4. Jan 2006 (CET)
richtig lesen freund ;) ist ja klar, dass das ausgangsmaterial aus zellen entnommen werden müssen. es wird jedoch die ganze praxis OHNE hilfe von bakterien in lustigen reagenzgläsern durchgeführt. halt eben nicht in vivo ;) und das ausgangsmaterial braucht man nur in geringen mengen, da es ja eben mit diesen verfahren repliziert wird. nicht so zwanghaft fehler suchen^^ hoffe ich habe dich nicht falsch verstanden
mfg MauZi
- Dann soll der Verfasser schreiben: "in vitro" ! --Wikipit 09:00, 5. Jan 2006 (CET)
Mangelhafte Lokalisierung
Eine hochwertige Enzyklopädie sollte sprachlich weitgehend lokalisiert sein. Die häufige Verwendung der engl. Abkürzung DNA (an Stelle von DNS) erscheint mir doch etwas gedankenlos. Sie gehört in die englische Fassung der Seite, die per Mausklick erreichbar ist. --Ribald 23:39, 4. Jan 2006 (CET)
Der Begriff DNA ist auch in der deutschen Wissenschaft gebrächlich. Die Verwendung englischsprachiger Begriffe bei jüngeren Erkenntnissen ist üblich. Allein die ABkürzung für die Polymerase-Kettenreaktion mit PCR und nicht PKR spricht dafür. (nicht signierter Beitrag von 84.191.84.213 (Diskussion) 1. Januar 2007)
Korrekturvorschlag zum Beispiel
Kleiner Hinweis: Man setzt für die PCR doch Desoxyribonukleotide ein, also dATP, dTTP, dGTP, dCTP. Bei den angegebenen Nukleotiden besteht die Gefahr der Verwechslung mit Ribobasen. Es gibt zwar Enzyme, die die ATP, GTP, UTP und CTP bei der PCR einbauen. Das ist aber nicht die Regel sondern ein seltener Spezialfall. --Maddy1971 23:42, 4.1.2006
Fehler in Artikel
Die Aussage das der gewünschte DNA Abschnitt in jedem Zyklus verdoppelt wird ist falsch! In den ersten beiden Zyklen erhalten wir nämlich ausschließlich Reaktionsprodukte die länger sind als der gewünschte Abschnitt.
->Das passiert auch in jedem folgendem Zyklus der PCR auch weiterhin! Ausserdem wird die Menge des Produktes nicht pro Zyklus genau verdoppelt, da die Amplifikationseffizienz selten 100% beträgt (gute Reaktionen liegen zwischen 90 und 100% also bei Amplifikationseffizienzen zwischen 1,9 und 2)! Ich finde aber, dass solche Kleinigkeiten dasVerständnis des Artikels erschweren würden! JBrain 12:44, 11. Jan 2006 (CET)
Meines Wissens ist für die Anlagerung der neue Nucleotide die Taq Polymerase verantwortlich
Habe diesen Fehler korrigiert allerdings nicht die im Text verwendeten Termini gebraucht. Das sollte noch verbessert werden.
Ergaenzungsvorschlaege
Im Abbild des Abschnittes "Ablauf" wuerde ich empfehlen, ganz unten bei der Benennung der einzelnen Ablaeufe, in klammern hinterzuschreiben, wie die eigentlichen Fachbegriffe lauten.
also unter
1 Schmelzen (Denaturierung)
2 Anlagerung(Hybritisierung)
3 Verlaengerung (Polymerisation)
und dass es sich bei der PCR-Methode um eine exponentielle Amplifizierung handelt, sollte meines Erachtens auch noch hervorgehoben werden werden... nur um das ewige Suchen nach der richtigen Terminologie der internetfaehigen Welt zu erleichtern...
Ich wiuerde es ja auch liebend gern selbst erledigen, allerdings verhindert mir dies die Editierungssperre..schade
herzlichen Gruß mic.user 22:01 , Feb 2006 (GMT+1)
- Deine Vorschläge habe ich umgesetzt -- danke! Ich weiß allerdings nicht wo man am besten exponentielle Amplifizierung unterbringt.
- Leider ist der Artikel halb-gesperrrt wie du es sagtest, finde ich sehr schade und ungerechtfertigt. Mal schaun was man da machen kann (infos hier: Wikipedia:Entsperrwünsche)
- Gruß --Saibo (Δ) 21:12, 7. Mär 2006 (CET)
- Update: Artikel ist entsperrt! --Saibo (Δ) 10:15, 9. Mär 2006 (CET)
Habe mal ein neues Bild aus Commons in den Artikel reingebaut, das zeigt die Schritte klarer und da ist auch die exponentielle Amplifizierung drin. Lanmar
Thermophile Organismen
Später wurde der PCR-Prozess verbessert, indem man die DNA-Polymerase von thermophilen Bakterien verwendete, die z.B. bei über 110 °C in Geysiren leben... Glaube, dass die keine 110°C aushalten! Meines Wissens ist die Grenze im Moment bei paarundneunzig °C bei Thermotoga spec. (bin aber kein Fachmann und lasse mich gerne eines Besseren belehren). Die Gattungen Thermus und Pyrococcus liegen knapp darunter. Die Proteine dieser Organismen sind z.T. bis über 110°C stabil (anderer AS Bias, immobilisierte Termini, stabiler faltende Cores, viele Chaperone etc.), unter diesen Bedingungen können die Organismen selber aber nicht überleben! Ausserdem kommen die nicht nur an Geysiren vor, sondern vor allem an Fumarolen wie z.B. Black smoker an MORs (Mittelozeanischen Rücken). Wäre nett, wenn das jemand der sich damit gut auskennt verbessern würde!--JBrain 11:31, 8. Mär 2006 (CET)
Genauigkeit
Wie hoch ist die statistische Genauigkeit der verwendeten Verfahren? 217.191.237.57 16:35, 24. Mär 2006 (CET)
Könntest Du diese Frage vielleicht etwas Präzisieren? Geht es um die Auswertung bei Vaterschaftstests oder gen. Fingerabdrücken oder um die PCR selber? Die verwendeten Enzyme machen ab und zu mal einen Fehler (Häufigkeit abhängig von den Reaktionsbedingungen und davon, ob sie eine 3'-5' Exonucleaseaktivität besitzen, auch 'Proofreading' genannt). Insgesamt aber ist es eine sehr genaue und sichere Methode! In der Phorensik ist die statistische Genauigkeit der Aussage (aus den Experimenten) meist nicht ein Problem der PCR selber, also der verwendeten Methode, sondern eher eine Verteilung der analysierten Marker in der Population! Die statistische Aussagekraft hängt also dabei von dem genauen Wissen über die Häufigkeit und Verteilung der einzelnen Marker in der menschlichen Population ab, nicht aber von der PCR selber. Ich hoffe, dass das die Frage einigermaßen beantwortet! Gruß --JBrain 17:05, 24. Mär 2006 (CET)
RNA
Im Artikel kommt der Begriff RNA nicht vor, es heißt eingangs, die PCR tauge zur Analyse der DNA. Nun sind aber Influenzaviren RNA-Viren, und (im Kontext von Influenza A/H5N1) wird immer wieder berichtet, dass deren Genom mit Hilfe der PCR analysiert wird. Im Artikel PCR müsste wohl ein kleiner Absatz stehen, wie das möglich ist: Muss die RNA erst in DNA umgeschrieben werden, oder taugt die PCR auch unmittelbar zur RNA-Analyse. Gruß: --Gerbil 15:26, 11. Apr 2006 (CEST)
- Das ist korrekt, RNA wird erst revers transkribiert und dann die PCR durchgeführt. Im Alltag wird das häufig in zwei getrennten Schritten gemacht, empfindlichere Verfahren beeinhalten eine Ein-Topf-Reaktion. Wie auch immer es läuft, das Ganze nennt sich RT-PCR. --Lode 15:53, 11. Apr 2006 (CEST)
PCR in der Praxis
Die eingschränkung der PCR auf Produktgrößen bis 1kb ist falsch. Während meiner Dipl.-Arbeit waren Produktgrößen bis 3kb kein Problem. Auch der aktuelle Beipackzettel der Taq-Polymerase von Roche Applied Science hat 3kb als max. Produktgröße angegeben. In diesem Bereich ist vor allem der Faktor Zeit während der Elongatuion wichtig (ca. 1 min je 1000 basen) Jiver 15:52, 7. Jun 2006 (CEST)
Richtig, und mit speziellen Long-Template-Kits ist noch mehr drin: über 20-40 kbp (z.B. v. Roche). --Jan R 13:17, 8. Jun 2006 (CEST)
Vorschlag für die Verbindung von "Ablauf" und "Schritte des PCR-Prozesses"
Da beide Absätze eindeutig das selbe beschreiben (noch dazu unterschiedliche Angaben machen), halte ich es für sinnvoll, sie zu einem Absatz zu verbinden. Hier ein Vorschlag: Jiver 10:35, 4. Jul 2006 (CEST)
Ablauf des PCR-Prozesses
Im Folgenden werden die einzelnen Schritte der PCR dargestellt. Alle gemachten Angaben sind als Richtwerte zu betrachten, die je nach Anforderung an die PCR angepasst werden müssen.
- Initialisierung (auch initiale Denaturierung). Das Gemisch wird ca. 2 Minuten lang auf 94-96 °C erhitzt, um sicherzustellen, dass sich die DNA-Doppelstränge getrennt haben. Die Wasserstoffbrückenbindungen, die die beiden DNA-Stränge zusammenhalten, werden aufgebrochen. Bei einer sogenannten “hot start-PCR” muss durch eine längere anfängliche Erhitzungs-Phase (bis zu 15 Minuten) die Polymerase erst freigesetzt bzw. aktiviert werden. Dieser Schritt wird einmalig vor der eigendlichen PCR-Reaktion durchgeführt um sicherzustellen, dass sich die doppelstängige Ausgangs-DNA als auch mögliche Primerdimere getrennt haben und als Einzelstränge vorliegen..
- Denaturierung (auch Melting, Schmelzen). Die Temperatur ca. 30 Sekunden lang auf 94-96 °C halten. Das genügt gewöhnlich, um die DNA während der Zyklen zu zerlegen.
- Annealing (auch Anlagerung, Primerhybridisierung). Die Temperatur ca. 30 Sekunden lang auf einer Temperatur halten, die eine spezifische Anlagerung der Primer an die DNA erlaubt. Meist liegt die Temperatur bei 50°C bis 65°C, die genaue Temperatur wird hierbei durch die Länge und die Anzahl komplementärer Nukleotide in der Primersequenz bestimmt (siehe Mutagenese). Wird die Temperatur zu niedrig gewählt, können sich die Primer u. U. auch an nicht 100%-komplementären Sequenzen anlagern und so zu unspezifischen Produkten (Geisterbanden) führen. Wird die Temperatur zu hoch gewählt, ist die thermische Bewegung der Primer u. U. so gross, dass sie sich nicht richtig anheften können, so dass es zu gar keiner oder nur ineffizienter Produktbildung kommt.
- [Elongation]] (auch Verlängerung, Polymerisation). Die Temperatur wird für ca. 30 Sekunden je 500 Basenpaare auf 68-72 °C (je nach verwendeter Polymerase) halten. Die DNA-Polymerase baut nun den fehlenden Stränge mit freien Nukleotiden auf. Sie beginnt dafür am 3'-Ende des Primers und folgt dann dem DNA-Strang, der als Matrize dient. Der Primer wird nicht wieder abgelöst, da er den Anfang des neuen Stranges bildet.Die Schritte 2-4 werden 25-40 mal wiederholt.
- Das Gemisch wird bei 4-8 °C aufbewahrt. Man kann die PCR am Abend vor dem Verlassen des Labors einleiten, so dass sie über Nacht läuft. Die DNA wird bei 4-8 °C nach nur einer Nacht nicht beschädigt.
Der Punkt 4 der Grafik ist nicht richtig, da sind nicht die alten Stränge wieder verbinden, sonder nach jedem Durchgang ein "neuer" und ein alter Strang miteinander verbunden sind
Aufreinigung von PCR-Produkten

Das PCR-Produkt kann durch Agarose-Gelelektrophorese anhand seiner Größe identifiziert werden. (Die Agarose-Gelelektrophorese ist ein Verfahren, bei der DNA in ein Agarose-Gel eingebracht wird und anschließend eine Spannung angelegt wird. Dann bewegen sich die kürzeren DNA-Stränge schneller als die längeren auf den Pluspol zu.) Die Menge des PCR-Produkts kann durch einen Vergleich mit einer DNA-Leiter, die DNA-Fragmente bekannter Größe enthält und parallel zur Probe im Gel mitläuft, bestimmt werden.
PCR bei HIV
Die PCR kann auch zu Reihenuntersuchungen eingesetzt werden. So wird sie z. B. von Blutspendediensten zur Routineuntersuchung von Blutkonserven eingesetzt. Durch die frühestmögliche Entdeckung einer gefährlichen Infektionskrankheit beim Spender (z. B. HIV, Hepatitis B) kann das sog. diagnostische Fenster nahezu geschlossen werden.
Kann obige Aussage seitens eines Fachmanns bezüglich HIV konkretisiert werden ? Ab welchen Zeitraum ( nach einem Risikokontakt ) kann man HIV mittels PCR nachweisen bzw. ausschliessen ? (nicht signierter Beitrag von 7. November 2006 (Diskussion | Beiträge) 195.200.34.50)
- Grundsätzlich sollte HIV durch einen Antikörpernachweis abgeklärt werden. Ca. 6 Monate nach HIV-Infektion ist es möglich mittels PCR den Virustiter zu bestimmen und die mittlere Zeit bis zum AIDS-Ausbruch zu prognostizieren. [1] Gruß Matthias 17:44, 22. Dez. 2020 (CET)
*Ketten*reaktion
Oma würde aus diesem Artikel gerne erfahren, warum dieser Prozess Kettenreaktion heißt. Wie wird da durch eine Einzelreaktion (d. h. die Reaktion von 1 Molekül) die nächste ausgelöst? --UvM 12:03, 7. Jul. 2008 (CEST)
- Weil der gesmate PCR-Prozess damit etwas beschreibt, was ähnlich wie eine Kettenreaktion abläuft. Man gibt die Proben in den Thermocycler, und der startet sozusagen eine Kettenreaktion, indem er die DNA-Stränge trennt, Primer ansetzt, Neue DNA Fragmente eibaut und so aus einem zwei, aus zwei vier, aus vier acht......... (theoretisch) unendnlich DNA verdoppelt (nat. mit Einschränkungen!). Aber du hast recht, würde man die Probe im Laborchargon "Vogelkakke" nennen, könnte man die PCR auch in "Vogelkakkevermehrung" umbenennen.^^ (nicht signierter Beitrag von 92.227.189.226 (Diskussion | Beiträge) 22:42, 18. Sep. 2009 (CEST))
Laser-assisted Nanodroplet PCR Technology
Hat jemand Zugriff zu obigem Artikel ? Entwickelt von SRI, 40 Zyklen in 6 Min., erschienen am 15. Jan. 2009. Gruss --Grey Geezer nil nisi bene 15:13, 16. Jan. 2009 (CET)
Test
wegen Wikipedia:Fragen zur Wikipedia#"Links auf diese Seite" sortiert?--HAL 9000 20:25, 2. Feb. 2009 (CET)
Weniger ist mehr
Ich würde gern die Anwendunggebiete Klonieren und Mutagenese unter Hinweis auf einen Hauptartikel etwas verschlanken. Einwände? -- Mr.crispy 13:41, 9. Jun. 2009 (CEST)
RT-PCR
RT-PCR steht im allgemeinen für Reverse Transkription und eigentlich nicht für "real time". Um hier Verwechslungen auszuschließen sollte die Abkürzung RT nur für die eigentliche Bedeutung Reverse Transkription verwendet werden. Ich denke dies sollte in dem Abschnitt klarer dargestellt werden. (nicht signierter Beitrag von 195.72.103.126 (Diskussion) 15:09, 20. Jun. 2011 (CEST))
Grundlegende Komponenten: Was ist mit der "Probe"?
Also die grundlegenden Komponenten (Template, Primer, Polymerase, etc.) werden im Abschnitt PCR in der Praxis sehr gut und nachvollziehbar erklärt (auch wenn man wie ich nicht vom Fach ist), nur vermisse ich eine Beschreibung dazu was im Englischen die "Probe" genannt wird. Könnte jemand mit Sachkenntnis dazu einen Satz (oder mehr) einfügen? Danke! -- 95.118.87.34 18:57, 21. Jun. 2011 (CEST)
Leichte Schwächen im Teil Quantitative Echtzeit-PCR (qRT-PCR)
Die beiden vorigen Kommentare beziehen sich auf diesen Abschnitt. Mir gefällt er so nicht besonders, weil er nur SYBR-Green beschreibt, als wäre es die einzige Variante. Taqmansonden (englisch taqman probes) sind aber auch sehr beliebt, und dann gibt es ja noch molecular beacons etc. Steht alles im speziellen Artikel Real time quantitative PCR, auf den ja auch schön verlinkt wird. M.E. wäre es besser, hier im Übersichtsartikel nur allerkürzestens zu erklären, dass pro Zyklus die Fluoreszenz gemessen wird und daraus auf die DNA-Menge geschlossen wird. Wo die Floureszenz herkommt, soll man im speziellen Artikel nachlesen. Auch mag ich die Überschrift nicht, man sollte das mit dem speziellen Artikel harmonisieren; RT-PCR bezeichnet tatsächlich v.a. reverse Transkription. Wenn Verwechslungen ausgeschlossen sind, kann man auch schon mal real-time meinen, aber in einem allgemeinen Artikel über die verschiedenen Sorten der PCR ist das nicht schön. Ich kann mich irgendwann mal drum kümmern, wenn jemand anders schneller ist, wär's mir auch recht... --Senfteiler 17:11, 10. Aug. 2011 (CEST)
Eineiige Zwillinge
Im Abschnitt PCR#Vaterschaftstest steht, dass bei eineiigen Zwillingen der genetische Fingerabdruck nahezu identisch sei. Eineiige Zwillinge haben doch aber per Definition das gleiche Genom und somit auch den gleichen genetischen Fingerabdruck? --Massenvektor 18:42, 29. Sep. 2011 (CEST)
- Bei der Zellteilung kommt es zu Mutationen.--84.41.34.154 00:51, 19. Jan. 2016 (CET)
- Beim Menschen sind es (größenordnungsmäßig) 200 Mutationen pro Generation. Das wäre wohl eine ein- oder zweistellige Zahl pro Zellteilung, auf 3 Milliarden Basen. Damit ist das Genom zwar technisch gesehen anders, aber die Chance, das auf einem PCR-basierten, genetischen Fingerabdruck zu sehen, geht gegen Null. Selbst bei kompletter Genom-Sequenzierung wäre das schwer nachzuweisen. Methylierungsmuster könnten da eher helfen. --Magnus Manske (Diskussion) 14:56, 19. Jan. 2016 (CET)
Theoretischer Ablauf - Widerspruch
Meiner Meinung nach widersprechen sich die Aussagen "Nach dem Schmelzen am Anfang des zweiten Zyklus stehen dadurch die beiden ursprünglichen DNA-Einzelstränge und zwei am 3’-Ende überlange Einzelstränge zur Verfügung." und "Im zweiten Zyklus stehen die eingesetzte DNA sowie die gerade gebildeten DNA-Stränge zur Verfügung. An Ersterer erfolgt derselbe Prozess wie im ersten Zyklus. An die neu gebildeten DNA-Einzelstränge, welche an 3’ bereits dort enden wo sie sollen, lagern sich nun wieder Primer in der 3’-Region an." . Die zu Beginn des 2. Zyklus zur Verfügung stehenden neu gebildeten Stränge besitzen (so wie im ersten Satz vermerkt) einen 3' Überhang und sind an ihrem 5' Ende bereits in der richtigen Länge ( aber nicht wie im 2. Satz des 2. Zitats vermerkt, an ihrem 3' Ende). Ich hoffe ich habe aber nicht nur einen Denkfehler... Gruß (nicht signierter Beitrag von 92.226.21.160 (Diskussion) 23:02, 8. Nov. 2011 (CET))
Theoretischer Ablauf - II
Ich habe ein Problem mit diesem Begriff. Ich weiss, was gemeint ist (als Gegensatz zur Praxis darüber), ABER man spricht auch nicht von dem Theoretischen Ablauf der DNA-Sequenzierung oder dem Theoretischen Ablauf der Proteinbiosynthese. Auch hier in dem Paragraph werden ganz konkrete Schritte und Reaktionen beschrieben. Worin besteht der Unterschied zwischen dem "theoretischen Ablauf" und dem "praktischen Ablauf"? Mir fehlt noch das richtige Wort, aber "theoretisch" erscheint mir hier im Zusammenhang zu abgehoben. Prinzipieller Ablauf? Abfolge der Reaktionen? Vorschläge? GEEZERnil nisi bene 11:40, 13. Jan. 2012 (CET) Im Engl. würde man sagen The Princile(s) of PCR.
- Jetzt, wo du es sagst... Wie wäre es mit "Prinzip der Methode"?--Senfteiler 18:04, 26. Feb. 2012 (CET)
- Stimme zu und habe es ganz simpel in "Prinzip" umbenannt. Erledigt. Enzoklop (Diskussion) 21:39, 14. Nov. 2020 (CET)
Q-PCR hat nur ein Beispiel und eine Referenz, dieses ist aber sehr detailliert
Das detaillierte Autreten nur eines Bsp. verzerrt die Wahrnehmung des Themas und riecht etwas nach Eigen- oder Bekanntenwerbung. -- Ghilt 13:43, 26. Feb. 2012 (CET)
- Vor allem geht es etwas am Thema des Artikels vorbei, es geht hier halt eigentlich um die sehr versatile Technik PCR und nicht um die jeweilige Lieblingsanwendung. Wenn unbedingt nötig, kann das ja im (übrigens sehr interessanten) Artikel über Real time quantitative PCR erörtert werden. Ich hatte ja schon vor einiger Zeit (10.8.11) hier angemerkt, dass der Abschnitt nicht ideal ist und evtl. deutlich gekürzt werden könnte. Mit einem Verweis auf den eigenen Artikel sollte das eigentlich konsensfähig sein, hoffe ich. Ich probier's mal: Beschränkung auf die Erklärung, dass Fluoreszenz während der Reaktion gemessen wird, und dass die mit der Menge der Amplikons korreliert. Dass die Fluoreszenz von verschiedenen Chemikalien kommen kann und schließlich noch eine Ermahnung, nicht RT zu sagen, wenn nicht reverse Transkription gemeint ist. Ich bin mal mutig. --Senfteiler 17:59, 26. Feb. 2012 (CET)
- Vielen Dank, --Ghilt 22:19, 26. Feb. 2012 (CET)
DNA - DNS, RNA - RNS
Da der Artikel in deutscher Sprache verfasst ist, sollten auch die deutschen Begriffe Eingang finden. Und im Deutschen heißt es nun mal "DesoxyriboNukleinSäure", also DNS. Analog bei RNS. (nicht signierter Beitrag von 84.169.196.66 (Diskussion) 22:00, 20. Dez. 2013 (CET))
Diese Frage ist bereits im 8. Abschnitt dieser Diskussion (“Mangelhafte Lokalisierung”) ausreichend beantwortet. Berni53 (Diskussion) 12:28, 8. Apr. 2015 (CEST)
Nobelpreis fehlt!
Zur Geschichte der PCR gehört meiner Meinung nach die Tatsache, dass sie 1993 mit dem Chemie-Nobelpreis für Mullis und Smith ausgezeichnet wurde! 134.247.251.245 12:19, 23. Aug. 2018 (CEST)
- Das stimmt. Deswegen ist das ja auch in der Einleitung zu finden. -- Oliver aus Hambergen Sprich! 12:20, 23. Aug. 2018 (CEST)
Video
Datei:Wie funktioniert der PCR-Test?.webm
Das nebenstehende Video wurde vom ZDF zur Verfügung gestellt. Da wir hier einen exzellenten Artikel haben möchte ich die Fachleute entscheiden lassen, ob das Video rein passt oder nicht. Angesicht des Abschnittes obendrüber würde er IMHO dem Artikel gut tun. -- sk (Diskussion) 21:23, 9. Nov. 2020 (CET)
- Die Idee finde ich prinzipiell gut und das Video erklärt das Prinzip der PCR auch recht anschaulich. Allerdings geht es im Video speziell um die Testung von SARS-CoV-2 mittels PCR. Da der PCR-Artikel aber die allgemeine Methode behandelt, ist das Video hier wahrscheinlich eher irreführend. Es entsteht der Eindruck, dass die PCR immer ein Test ist und ein positives oder negatives Ergebnis liefert. Das Video wurde jedoch im Abschnitt RT-PCR im Artikel SARS-CoV-2 eingebettet. Dort schafft es einen großen Mehrwert. Enzoklop (Diskussion) 23:32, 19. Dez. 2020 (CET)
Corona
Guten Tag!
Über den PCR-Test, der scheinbar auch zur Feststellung eine Corona-Infektion (bin medizinischer Leie) verwendet wird, habe ich den Artikel hier gefunden. Leider steht hier nichts im Bezug zum Erkennen von Viren. Ist dieser Test denn sicher beim Auffinden von Virusinfektionen oder ist dieser Test in diesem Fall fehleranfällig? Schön wäre hierzu ein Abschnitt bezüglich Viren, da wohl nicht nur bei CoVid19 (hoffentlich stimmt das) dieser Test angewendet wird. Warum dauert es zum Teil mehrere Arbeitstage, bis das Ergebnis vorliegt und im anderen Fall nur zwei Tage? (nicht signierter Beitrag von 2A03:F580:84BC:B200:4175:37C9:8187:18EE (Diskussion) 22:12, 7. Apr. 2020 (CEST))
- Die PCR eignet sich zum Nachweis jeglicher DNA-Sequenzen, mit einer vorangehenden reversen Transkription auch für RNA. Diagnostische PCR werden methodisch validiert. Der Test dauert mit Extraktion und RT und PCR max. 4-5 Stunden. Der Rest der Zeit ist Logistik. --Ghilt (Diskussion) 22:30, 7. Apr. 2020 (CEST)
- Ein befreundeter Unfallchirurg sagte mir neulich, das Laboratorium in seinem Krankenhaus könne ihm bereits nach 2-3 Stunden ein Ergebnis liefern. (Davon hängt ab, welche Schutzkleidung er tragen muß.) Diese Aussage ist natürlich OR und somit nicht direkt für den Artikel verwertbar, aber vielleicht hat ja jemand auch vernünftige Quellen für die minimale Zeit zur Hand. --Bogdan Wolynetz (Diskussion) 15:55, 23. Aug. 2020 (CEST)
Die Vervielfältigung liefert lediglich ein 'mehr' an DNS. Wie wird der Abgleich auf das gesuchte Objekt ausgeführt? (COVID, Influenza, Mörder-vom-dunklen-Wald) Eventuell paßt das nicht mehr zum Thema, aber ein Link wäre nett. (nicht signierter Beitrag von 178.14.20.106 (Diskussion) 16:19, 23. Sep. 2020 (CEST))
Hallo,ist es möglich durch Veränderung des PCR Tests(z.B.Zyklen)mehr oder auch weniger positive oder negative Testergebnisse zu erzeugen? (nicht signierter Beitrag von 2A02:B98:4731:74:9D73:EB:81C0:CFD7 (Diskussion) 18:28, 14. Nov. 2020 (CET))
- Lässt man die PCR länger laufen (mehr Zyklen) so kann man die Empfindlichkeit erhöhen. Bei der Validierung eines PCR-Tests wird die geeignete Zyklenzahl ermittelt. Dies kann abhängig vom Gerät oder von den Reagenzien sein und ist oft von Hersteller zu Hersteller unterschiedlich. Ziel ist es das richtige Ergebnis zu ermitteln und nicht "mehr oder weniger" positive Testergebnisse zu "erzeugen". Die Maschine würfelt nicht, sondern kann exakt definierte DNA-Abschnitte mit einer gewissen Nachweisgrenze anzeigen. Gruß Matthias 17:27, 22. Dez. 2020 (CET)
- Kommt drauf was man mit den Tests beabsichtig. Mehr Zyklen = Hoehere Wahrscheinlichkeit auch Falsche Positive zu bekommen. Dass kann durchaus erwuenscht sein, wenn man sich davon einen Nutzen verspricht. --105.8.0.178 19:26, 12. Jan. 2021 (CET)
Hallo, der Kontext, mit dem ich hier gerade unterwegs bin, besteht darin, dass ein Freund bzgl. COVID-19 sehr verschwörungsgläubig geworden ist und ich mit ihm recht scharfe Auseinandersetzungen hatte. Außerdem bin ich ein totaler Laie bzgl. PCR-Tests. Aus diesem Kontext heraus ergeben sich für mich beim aktuellen Stand des Artikels die folgenden Fragen:
- Liegt bei Durchführung einer PCR die Anzahl der Zyklen schon vorher fest? Wenn ja, woraus bestimmt sich die Zahl? Und muss ich im negativen Fall den Test unter bestimmten Umständen mit höherer Zyklenzahl wiederholen? Vielleicht mit einem anderen Test, dessen empfohlene Zyklenzahl höher liegt? Matthias hatte weiter oben bereits einige Andeutungen dazu gemacht, mich würde dieses Thema im Detail interessieren. Außerdem scheint im Falle von quantitativen Echtzeit-PCRs die Zyklenzahl wohl variabel zu sein. Ein wenig was dazu steht unter Real_Time_Quantitative_PCR#Ct-Wert_oder_auch_Cp-Wert; sollte der Artikel um einen Abschnitt zu dieser Frage ergänzt werden, wäre es vielleicht sinnvoll, auch darauf zu verweisen.
- Sind die meisten durchgeführten Tests zum Nachweis von SARS-CoV-2 nicht nur RT-PCRs, sondern sogar qRT-PCRs (also auch Quantitative Echtzeit PCRs)? Ah, ich sehe gerade, dass diese Frage unter SARS-CoV-2#RT-PCR-Test positiv beantwortet wird. Gut, das ist eine Frage, die dieser Artikel hier wohl auch nicht beantworten muss.
- Theoretisch würde ja ein einziger DNA-Strang (bzw. RNA-Strang im Falle einer RT-PCR) genügen, um mit der PCR-Methode beliebig viele Kopien von primer-definierten Abschnitten zu erzeugen. Am Ende des Abschnitts "Ablauf" heißt es aber: "Dies ist der theoretische Idealfall, in der Praxis fallen aber zudem in geringem Maße auch kürzere Fragmente als die gewünschte Ziel-DNA an. Diese kurzen Fragmente häufen sich vor allem in den späten Zyklen an und können durch Fehlpaarung der Primer auch zu falschen PCR Produkten werden. Daher werden bei PCR Reaktionen meist nur etwa 30 Zyklen durchlaufen, damit vorwiegend DNA der gewünschten Länge und Sequenz produziert wird." Dies limitiert also Methode dahingehend, dass die Vervielfältigung nicht beliebig weit getrieben werden kann. Daher die Frage: Gibt es Umstände, unter denen mehr als eine Startsequenz benötigt wird, damit ein klar erkennbares positives Ergebnis überhaupt zustande kommen kann? Falls ja, würden mich Beispiele interessieren. Auch hier gibt es Aussagen unter Real_Time_Quantitative_PCR#Ct-Wert_oder_auch_Cp-Wert, die wohl etwas zur Beantwortung dieser Frage beitragen, aber reichen tun die mir nicht. Die Frage ist für mich deswegen so wichtig, weil ich so exakt wie möglich bestimmen möchte, was ein positives bzw. negatives Ergebnis eines PCR-Tests zum Nachweis von SARS-CoV-2 aussagt. Es ist etwas anderes, ob so ein Test die Anwesenheit bestimmter RNA-Fragmente (also auch eines einzelnen Fragments) ja/nein beantwortet, oder ob der Test beantwortet, ob die Menge bestimmter RNA-Fragmente oberhalb eines bestimmten Schwellwerts liegt oder nicht. Das kann doch möglichwerweise juristisch ebenso wie epidemiologisch relevant sein; in jedem Fall ist aber ein korrektes Verständnis der Ergebnisse dieser Tests vonnöten. Ein Abschnitt zu dieser Frage in diesem Artikel erscheint mir sinnvoll und wünschenswert, ggf. unter Auslagerung sehr spezifischer Infos in den SARS-CoV-2-Artikel.
--Dissenswurst (Diskussion) 13:05, 19. Feb. 2021 (CET)
Kritik
Das Wort Kritik taucht kein einziges Mal im Artikel auf. Gibt es im Deutschland des Jahres 2020 nur noch die Regierungsmeinung? - https://www.achgut.com/artikel/corona_politik_grundlegende_pcr_test_studie_auf_treibsand_gebaut (nicht signierter Beitrag von 2003:E6:1F03:9901:94C3:A715:9219:800E (Diskussion) 11:14, 2. Dez. 2020 (CET))
- Kritik an der Polymerase-Kettenreaktion von "achgut"? Wäre witzig. Du willst wahrscheinlich irgendwas über Covid / Corona sagen. Aber das "taucht kein einziges Mal im Artikel auf". --Logo 11:32, 2. Dez. 2020 (CET)
- Der Artikel von achgut bezieht sich auf das hier: https://cormandrostenreview.com/report/
- Das ist eine massive Kritik von Wissenschaftlern an dem ursprünglichen Artikel von Corman, Drosten und anderen vom 23.01.2020 in Eurosurveillanvce zur Entwicklung eines PCR-Tests zum Nachweis von SARS-COV-2 (https://doi.org/10.2807/1560-7917.ES.2020.25.3.2000045). Diese Kritik wurde auch bei Eurosurveillance eingereicht. Eurosurveillance hat darauf inzwischen geantwortet. Ich will hier nicht weiter darauf eingehen, denn als PCR-Laie kann ich zu etlichen Punkten, die in dieser Kritik aufgeworfen wurden, nichts sagen, und die Antwort von Eurosurveillance geht leider nicht im Detail auf alle aufgeworfenen Punkte ein. Die Wikipedia könnte diesen Vorgang durchaus erwähnen, ich denke aber, dass diese Kritik ja nicht die PCR-Methode allgemein betrifft, sondern speziell den Test zum Nachweis von SARS-CoV-2 und daher im Artikel SARS-CoV-2 besser aufgehoben wäre. Die Arbeit von Corman et al. wird dort auch massiv angeführt (Einzelnachweis Nr. 31.), daher könnte man Kritik daran und Reakionen darauf sicher auch erwähnen. Im FOCUS z.B. wird Jonas Schmidt-Chanasit zitiert, der der die oben erwähnte Kritik als eine "Desinformationskampagne" wertet (https://www.focus.de/gesundheit/news/internationale-forscher-stecken-hinter-papier-virologe-schmidt-chanasit-haerte-kann-man-nicht-einfach-mit-wirksamkeit-gleichsetzen_id_12752954.html). Leider geht der Focus-Aritkel nur auf sehr wenige Punkte ein; für Laien wäre eine detaillierte Auseinandersetzung mit der aufgeworfenen Kritik interessant. Es mag aber nicht die Aufgabe der Wikipedia sein, hier allzusehr in die Einzelheiten zu gehen. --Dissenswurst (Diskussion) 11:47, 5. Mär. 2021 (CET)
Frage zu Schmelzpunkten
Ich bin totaler PCR-Laie. Frage: Zunächst wird erhitzt, bis es zur Schmelze der DNA kommt. Wenn nun wieder abgekühlt wird, warum renaturiert die DNA nicht sofort wieder (so dass die Primer sich gar nicht anlagern könnten)? Dafür gibt es sicher einen guten Grund, und es wäre schön, wenn der Artikel das klären würde. Ich hätte aufgrund dieser Vorstellung erwartet, dass die Anlagerung der Primer bei einer Temperatur stattfindet, die über dem Schmelzpunkt der DNA liegt. Im Artikel zur DNA heißt es beim Thema Schmelzpunkt akuell (siehe Desoxyribonukleinsäure#Schmelzpunkt, letzter Absatz): "Wenn die Temperatur der Lösung unter Tm zurückfällt, können sich die Einzelstränge wieder aneinanderlagern. Dieser Vorgang heißt Renaturierung oder Hybridisierung." Leider ist hier nicht genau ausgeführt, ob das sofort passiert oder dieser Vorgang eine gewisse Trägheit aufweist. Das Wort "können" lässt sogar offen, ob es überhaupt zwangsläufig oder nur mit einer gewissen Wahrscheinlichkeit passiert (und wie das möglicherweise damit zusammenhängt, wie weit die Temperatur wieder unter Tm sinkt). --Dissenswurst (Diskussion) 11:15, 5. Mär. 2021 (CET)
- Erstmal: Die Anlagerung findet per Definition unterhalb eines Schmelzpunktes statt (überhalb ist es ja "geschmolzen"). Dann ist es wichtig auseinanderzuhalten, dass es zwei relevante Schmelzpunkte gibt. Einmal von Template/Template und dann von Primer/Template. Wenn man im Kontext von PCR von Tm spricht, dann ist eigentlich immer die Schmelztemperatur des Primers gemeint. Zur Berechnung gibt es eine Formel, es gilt: je länger die Sequenz, desto höher die Temperatur. Während man im ersten Schritt (Denaturierung) pauschal 95°C wählt, muss die Annealing-Temperatur spezifisch für den verwendeten Primer gewählt werden. Man rechnet dazu erst Tm von Primer/Template aus und die Annealing-Temperatur, bei der die Primer sich gerade noch so, bzw. schon, anlagern, ist dann 5–10°C darunter. Warum legt sich das Template dann nicht schon vor dem Primer an und verhindert dadurch eine Anlagerung der Primer? Die Primer sind wesentlich kürzer und in viel höheren Konzentrationen vorhanden. Sie finden einfach schneller das komplementäre Gegenstück. --Enzoklop (Diskussion) 18:12, 10. Apr. 2021 (CEST)