Metabolitenschaden
Unter Metabolitenschäden versteht man Schäden, die an Metaboliten entstehen. Ein Metabolitenschaden kann durch Nebenreaktionen von Enzymen oder spontane chemische Reaktionen auftreten. Viele Metaboliten sind chemisch reaktiv und instabil und können mit anderen zellulären Komponenten reagieren oder anderweitig chemisch umgewandelt werden.
Schädigungsarten
Ähnlich wie DNA und Proteine, sind auch Metaboliten anfällig für unerwünschte Nebenreaktionen, deren Reaktionsprodukte als Beschädigungen bezeichnet werden. Beschädigungen können chemisch oder enzymatisch durch enzymatische Nebenreaktionen passieren. Während DNA- und Protein-Schäden relativ gut erforscht sind, ist Metabolitenschädigung ein neues Forschungsfeld. Dies liegt unter anderem auch an der hohen Vielfalt und Anzahl an reaktiven (und damit schadensanfälligen) Metaboliten.
Chemische Schädigung
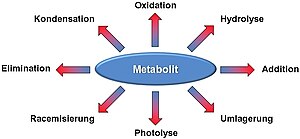
Viele Metaboliten sind chemisch reaktiv und instabil und daher anfälliger für chemische Schädigung. Im Allgemeinen kann jede chemische Reaktion, die außerhalb eines lebenden Organismus (in vitro) unter physiologischen Bedingungen stattfindet, auch in einem lebendigen Organismus (in vivo) stattfinden.[1][2] Typische Arten von chemischen Schädigungen an Metaboliten, die in der Zelle vorkommen können, sind Racemisierung, Umlagerung, Eliminierung, Photolyse, Addition und Kondensation.
Einige Metaboliten sind so reaktiv, dass ihre Halbwertszeit in Minuten gemessen wird. Zum Beispiel hat 1,3-Bisphosphoglycerat, ein Zwischenprodukt der Glykolyse, eine Halbwertszeit von 27 Minuten in vivo.[3]
Enzymatische Schädigung
Obwohl Enzyme im Allgemeinen eine hohe Substratspezifität haben, können Nebenreaktionen von Enzymen zu unbrauchbaren, zum Teil auch giftigen Produkten führen. Die Reaktionsgeschwindigkeiten dieser Nebenreaktionen sind zwar wesentlich langsamer als die der metabolischen Reaktionen, dennoch kann die Anhäufung von beschädigten Metaboliten (Nebenprodukten) im Laufe der Zeit jedoch bedeutsam sein. Zum Beispiel reduziert die mitochondriale Malatdehydrogenase das Substrat α-Ketoglutarsäure zu L-2-Hydroxyglutarat 107 mal langsamer als das eigentliche Substrat Oxalacetat. L-2-Hydroxyglutarat kann dennoch zu mehreren Gramm pro Tag im erwachsenen Menschen anfallen.[4]
Schadenskontrollsysteme
Beschädigte Metaboliten (durch enzymatische oder chemische Beschädigung) haben einen veränderten, meist geringeren Nutzen in der Zelle und sind oftmals giftig. Um zelluläre Vergiftungen zu vermeiden, die durch Anhäufung von beschädigten Metaboliten auftreten können, haben Organismen Mechanismen entwickelt. Diese Schadenkontrollsysteme können ein oder mehrere spezifische Enzyme umfassen.[5][6] Eine Schädigung an Makromolekülen durch spontane Reaktionen oder enzymatische Fehler kann durch enzymatische Kontrollsysteme repariert werden. Das gleiche Prinzip gilt auch für Metabolite die anfällig für Schädigung durch spontane chemische Reaktionen oder enzymatische Fehler sind. Metaboliten Schadenskontrollsysteme können entweder den Schaden direkt reparieren (Reparatur) oder ihn verhindern (Prävention). Ein gelenkter Überlauf ist ein Spezialfall der Prävention. Es werden drei Kategorien von Schadenskontrollsystemen unterschieden:
Reparatur von beschädigten Metaboliten
Die Reparatur beinhaltet die Umwandlung von beschädigten Metaboliten zurück zu deren ursprünglichen Zuständen. Das Konzept ist ähnlich zur DNA-Reparatur. Zum Beispiel reduziert die Malatdehydrogenase in einer Nebenreaktion α-ketoglutarat zu L-2-hydroxyglutarat. Dieses Produkt stellt eine Sackgasse im Metabolismus dar und ist kein Substrat für andere Enzyme im Zentralmetabolismus. Eine Anhäufung von L-2-hydroxyglutarat führt außerdem zu L-2-hydroxyglutaric aciduria, einer Stoffwechselkrankheit. Das Reparaturenzym α-Hydroxyglutaratdehydrogenase oxidiert L-2-hydroxyglutarat zurück zu α-ketoglutarat, und repariert somit das Metabolit.[4]
Prävention von Metabolitenschädigung
Prävention unterbindet zukünftige Schäden, bevor sie passieren können. Zwei Mechanismen in der Prävention werden unterschieden: Zum Einen können reaktive Metaboliten zu weniger schädlichen Metaboliten umgewandelt werden, oder physiologische, aber langsame Reaktionen werden enzymatisch beschleunigt. Die reaktiven Metaboliten können dabei entweder Nebenprodukte sein, oder normale, aber hochreaktive Zwischenstufen im Zentralmetabolismus.
Eine Nebenreaktion der Rubisco führt beispielsweise zu geringen Mengen an Xylulose-1,5-Bisphosphat, welches die Rubisco-Aktivität inhibiert. Das Enzym CbbY dephosphoryliert Xylulose-1,5-Bisphosphat zu dem natürlichen Metabolit Xylulose-5-Phosphat, und verhindert damit die Hemmung der Rubisco Aktivität.[7]
Gelenkter Überlauf
Der gelenkte Überlauf ist ein Spezialfall der Prävention, bei dem ein Überschuss von physiologischen, aber reaktiven Metaboliten zu giftigen Produkten führen kann. Verhindern dieses Überschusses ist daher Präemption von potenziellen Schädigungen.
Die ersten zwei Zwischenprodukte in der Biosynthese von Riboflavin sind extrem reaktiv und können spontan zu 5-Phosphoribosylamin und Maillard-Reaktionsprodukten zerfallen, die ihrerseits hochreaktiv und schädlich sind. Das Enzym COG3236 spaltet diese ersten beiden Zwischenprodukte in weniger schädliche Produkte und verhindert damit mögliche Schädigungen, die andererseits entstehen könnten.[8]
Metabolitenschädigung und Krankheiten
L-2-Hydroxyglutaric aciduria war die erste menschliche Krankheit, die mit einem fehlenden Reparaturenzym für Metaboliten in Verbindung gebracht wurde. Eine Mutation im L2HGDH Gen führt zur Ansammlung von α-Hydroxyglutarat, welches strukturell Glutamat und α-Ketoglutarat ähnelt, und dadurch wahrscheinlich andere Enzyme und Transporter inhibiert.[4]
Systembiologie
Modellierungen in der Systembiologie zielen darauf ab, den zellulären Metabolismus in silico zu reproduzieren. Metabolitenschädigung und deren Reparatur verbrauchen einen Anteil der zellulären Energiekapazitäten und müssen daher in diese Modelle eingearbeitet werden um „Metabolic Engineering“-Projekte besser anzuleiten.[5]
Außerdem können Metaboliten Reparaturenzyme für einen großen Teil der unbekannten, konservierten Gene kodieren, die es in allen bisher sequenzierten Organismen gibt.[5][6]
Synthetische Biologie / „Metabolic Engineering“
Bei der Konstruktion von Stoffwechselwegen in Organismen, aber auch wenn ein nativer Stoffwechselweg massiv hochreguliert wird ("metabolic engineering"), können reaktive Stoffwechselzwischenprodukte anfallen und den Wirtsorganismus negativ beeinträchtigen, weil der zuständige Reparaturweg fehlt oder dessen Durchflussrate nicht angepasst ist.[9] Die Konstruktion von Schadenskontrollsystemen kann daher in der synthetischen Biologie und im „Metabolic Engineering“ notwendig sein.[10]
Einzelnachweise
- ↑ A. G. Golubev: [The other side of metabolism]. In: Biokhimija (Moscow, Russia). Band 61, Nr. 11, 1996, ISSN 0320-9725, S. 2018–2039, PMID 9004862.
- ↑ Markus A. Keller, Gabriel Piedrafita, Markus Ralser: The widespread role of non-enzymatic reactions in cellular metabolism. In: Current Opinion in Biotechnology. Band 34, 2015, ISSN 1879-0429, S. 153–161, doi:10.1016/j.copbio.2014.12.020, PMID 25617827, PMC 4728180 (freier Volltext).
- ↑ Erwin Negelein: [36] Synthesis, determination, analysis, and properties of 1,3-diphosphoglyceric acid. Band 3. Academic Press, 1. Januar 1957, S. 216–220 (sciencedirect.com [abgerufen am 23. September 2016]).
- ↑ a b c E. Van Schaftingen, R. Rzem, M. Veiga-da-Cunha: L-2-Hydroxyglutaric aciduria, a disorder of metabolite repair. In: Journal of Inherited Metabolic Disease. Band 32, Nr. 2, 2009, ISSN 1573-2665, S. 135–142, doi:10.1007/s10545-008-1042-3, PMID 19020988.
- ↑ a b c Carole L. Linster, Emile Van Schaftingen, Andrew D. Hanson: Metabolite damage and its repair or pre-emption. In: Nature Chemical Biology. Band 9, Nr. 2, 2013, ISSN 1552-4469, S. 72–80, doi:10.1038/nchembio.1141, PMID 23334546.
- ↑ a b Andrew D. Hanson, Christopher S. Henry, Oliver Fiehn, Valérie de Crécy-Lagard: Metabolite Damage and Metabolite Damage Control in Plants. In: Annual Review of Plant Biology. Band 67, 2016, ISSN 1545-2123, S. 131–152, doi:10.1146/annurev-arplant-043015-111648, PMID 26667673.
- ↑ Andreas Bracher, Anurag Sharma, Amanda Starling-Windhof, F. Ulrich Hartl, Manajit Hayer-Hartl: Degradation of potent Rubisco inhibitor by selective sugar phosphatase. In: Nature Plants. Band 1, 2015, ISSN 2055-0278, S. 14002, doi:10.1038/nplants.2014.2, PMID 27246049.
- ↑ Océane Frelin, Lili Huang, Ghulam Hasnain, James G. Jeffryes, Michael J. Ziemak: A directed-overflow and damage-control N-glycosidase in riboflavin biosynthesis. In: The Biochemical Journal. Band 466, Nr. 1, 2015, ISSN 1470-8728, S. 137–145, doi:10.1042/BJ20141237, PMID 25431972, PMC 4477702 (freier Volltext).
- ↑ Vincent J. J. Martin, Douglas J. Pitera, Sydnor T. Withers, Jack D. Newman, Jay D. Keasling: Engineering a mevalonate pathway in Escherichia coli for production of terpenoids. In: Nature Biotechnology. Band 21, Nr. 7, 2003, ISSN 1087-0156, S. 796–802, doi:10.1038/nbt833, PMID 12778056.
- ↑ Sydnor T. Withers, Shayin S. Gottlieb, Bonny Lieu, Jack D. Newman, Jay D. Keasling: Identification of isopentenol biosynthetic genes from Bacillus subtilis by a screening method based on isoprenoid precursor toxicity. In: Applied and Environmental Microbiology. Band 73, Nr. 19, 2007, ISSN 0099-2240, S. 6277–6283, doi:10.1128/AEM.00861-07, PMID 17693564, PMC 2075014 (freier Volltext).