Pyruvat-Phosphat-Dikinase
| Pyruvat-Phosphat-Dikinase | ||
|---|---|---|

| ||
| Struktur der PPDK aus Zea mays (PDB 1VBH). Farblich abgesetzt sind die Nukleotid-Bindedomäne (grün), PEP/Pyruvat-Bindedomäne (blau) mit dem Substrat PEP und einem Mg2+-Ion, die Phosphohistidin-Domäne (gelb), sowie die Linker-Peptide (rot). Der katalytische Histidin-Rest ist durch einen Pfeil hervorgehoben. Die Überlagerung (grau) zeigt die PPDK aus Clostridium symbiosum (PDB 1KBL). | ||
| Masse/Länge Primärstruktur | 947 Aminosäuren (Zea mays) | |
| Sekundär- bis Quartärstruktur | Homotetramer | |
| Kofaktor | Mg2+, NH4+ | |
| Bezeichner | ||
| Gen-Name(n) | pdk (MaizeGDB) | |
| Externe IDs |
| |
| Enzymklassifikation | ||
| EC, Kategorie | 2.7.9.1, Phosphotransferase | |
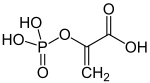
| Substrat | ATP + Pyruvat + Phosphat | |
| Produkte | AMP + Phosphoenolpyruvat + Diphosphat | |
| Vorkommen | ||
| Übergeordnetes Taxon | Pflanzen, div. Bakterien und Protozoen | |
Die Pyruvat-Phosphat-Dikinase (PPDK) (EC 2.7.9.1) gehört zur Enzymklasse der Phoshphotransferasen und katalysiert die ATP-abhängige Phosphorylierung von Pyruvat zu Phosphoenolpyruvat.
Erstmals beschrieben wurde die PPDK in Gräsern[1] und der parasitär lebenden Amöbe Entamoeba histolytica.[2] Einige hauptsächlich anaerob lebende Bakterien und Protozoen nutzen diese Reaktion in umgekehrter, Pyruvat formender Richtung zur Gewinnung von ATP.[3]
Katalysierte Reaktion
ATP, Pyruvat und Phosphat werden zu AMP, Phosphoenolpyruvat (PEP) und Diphosphat umgesetzt.
Strukturelle Funktionalität
Unter Berücksichtigung von Sequenzhomologien und Strukturdaten kann die PPDK in drei Domänen unterteilt werden:
- Nukleotid-Bindedomäne
- Zentrale Phosphohistidin-Domäne
- PEP/Pyruvat-Bindedomäne
Innerhalb der Nukleotid-Bindedomäne formen 240 Aminosäurereste eine ATP-grasp, 340 Reste der C-terminalen PEP/Pyruvat-Bindedomäne bilden ein so genanntes TIM-Fass.[4] Die drei Domänen sind über flexible Linker-Peptide miteinander verbunden.
Der Abstand zwischen der Nukleotid- und der PEP/Pyruvat-Bindedomäne beträgt etwa 45 Å. Eine direkte Interaktion beider Substratbindedomänen ist über diese Distanz nicht möglich. Ermöglicht wird die Phosphatübertragung stattdessen über eine Torsionsbewegung der Phosphohistidin-Domäne.[5]
Funktion in C4-Pflanzen
In C4-Pflanzen ist die PPDK in den Chloroplasten der Mesophyllzellen lokalisiert und katalysiert dort die Regeneration des primären CO2-Akzeptors Phosphoenolpyruvat.
Literatur
- C. J. Chastain, C. J. Failing, L. Manandhar, M. Zimmerman, M. M. Lakner, T. H. T. Nguyen: Functional evolution of C4 pyruvate, orthophosphate dikinase. In: Journal of Experimental Botany. Band 62, Nr. 9, 17. Mai 2011, S. 3083–3091, doi:10.1093/jxb/err058, PMID 21414960.
- A. S. Raghavendra, R. F. Sage (Hrsg.): C4 Photosynthesis and Related CO2 Concentrating Mechanisms (= Advances in Photosynthesis and Respiration. Band 32). Springer Netherlands, Dordrecht 2011, ISBN 978-90-481-9406-3, doi:10.1007/978-90-481-9407-0.
Einzelnachweise
- ↑ M. D. Hatch, C. R. Slack: A new enzyme for the interconversion of pyruvate and phosphopyruvate and its role in the C4 dicarboxylic acid pathway of photosynthesis. In: The Biochemical Journal. Band 106, Nr. 1, Januar 1968, S. 141–146, doi:10.1042/bj1060141, PMID 4305612, PMC 1198479 (freier Volltext).
- ↑ R. E. Reeves: A new enzyme with the glycolytic function of pyruvate kinase. In: The Journal of Biological Chemistry. Band 243, Nr. 11, 10. Juni 1968, S. 3202–3204, PMID 4297474.
- ↑ D. J. Pocalyko, L. J. Carroll, B. M. Martin, P. C. Babbitt, D. Dunaway-Mariano: Analysis of sequence homologies in plant and bacterial pyruvate phosphate dikinase, enzyme I of the bacterial phosphoenolpyruvate: sugar phosphotransferase system and other PEP-utilizing enzymes. Identification of potential catalytic and regulatory motifs. In: Biochemistry. Band 29, Nr. 48, 4. Dezember 1990, S. 10757–10765, doi:10.1021/bi00500a006, PMID 2176881 (englisch).
- ↑ O. Herzberg, C. C. Chen, S. Liu, A. Tempczyk, A. Howard, M. Wei, D. Ye, D. Dunaway-Mariano: Pyruvate Site of Pyruvate Phosphate Dikinase: Crystal Structure of the Enzyme−Phosphonopyruvate Complex, and Mutant Analysis. In: Biochemistry. Band 41, Nr. 3, 22. Januar 2002, S. 780–787, doi:10.1021/bi011799+, PMID 11790099.
- ↑ O. Herzberg, C. C. Chen, G. Kapadia, M. McGuire, L. J. Carroll, S. J. Noh, D. Dunaway-Mariano: Swiveling-domain mechanism for enzymatic phosphotransfer between remote reaction sites. In: Proceedings of the National Academy of Sciences of the United States of America. Band 93, Nr. 7, 2. April 1996, S. 2652–2657, doi:10.1073/pnas.93.7.2652, PMID 8610096, PMC 39685 (freier Volltext).