Assemblin
| Dimeres Assemblin | ||
|---|---|---|

| ||
| Dimeres Pseudorabies-Virus-Assemblin (ΔAla225), inhibiert mit DFP (Kalottenmodell). Gestrichelte Linie: Dimerachse, violett: katalytische Triade, cyan: oxyanion-hole loop, nach PDB 4V08 | ||
|
Vorhandene Strukturdaten: 4V07, 4V08, 1CMV, 1VZV, 1AT3, 1O6E, 1FL1, 2PBK | ||
| Masse/Länge Primärstruktur | 24,5 kDa bzw. 225 Aminosäuren je Monomer (pseudorabies virus) | |
| Sekundär- bis Quartärstruktur | Homodimer | |
| Bezeichner | ||
| Gen-Name(n) | UL26 | |
| Externe IDs | ||
| Enzymklassifikation | ||
| EC, Kategorie | 3.4.21.97, Serinprotease | |
| MEROPS | S21.001 | |
| Reaktionsart | Hydrolyse | |
| Substrat | a) assembly protein b) Gerüstprotein | |
| Produkte | a) Assemblin, Gerüstprotein mit Linker, reifes Gerüstprotein mit Linker, Hauptkapsidprotein-Bindungsstelle b) reifes Gerüstprotein, Hauptkapsidprotein-Bindungsstelle | |
| Vorkommen | ||
| Übergeordnetes Taxon | Herpesvirales | |
| Orthologe | ||
| HSV-1 | HCMV | |
| Entrez | 2703453 | 3077485 |
| UniProt | P10210 | P16753 |
| Refseq (Protein) | YP_009137100.1 | YP_081529.1 |
| PubMed-Suche | 2703453 | 3077485
|
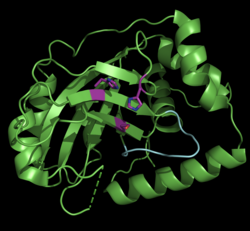
| Monomeres Assemblin | ||
|---|---|---|
| ||
| Monomeres Pseudorabies-Virus-Assemblin (ΔAla225), violett: katalytische Triade, cyan: oxyanion-hole loop, nach PDB 4V0T (Kette A) | ||
|
Vorhandene Strukturdaten: 4V0T | ||
| Masse/Länge Primärstruktur | 24,5 kDa, 225 Aminosäuren (pseudorabies virus) | |
| Sekundär- bis Quartärstruktur | Monomer | |
Assembline sind eine Klasse von Serinproteasen, die in allen Herpesviren vorkommen und eine einzigartige Katalytische Triade haben. Diese besteht aus einem Serin und zwei Histidinen. Die Aktivität der Assembline ist für die Virusreplikation essentiell und wird durch ein Monomer-Dimer-Gleichgewicht reguliert. Das schwach assoziierte Dimer (KD im mikromolaren Bereich) ist dabei die aktive Spezies. Die Gleichgewichtslage ist abhängig von der Assemblin- und der Salzkonzentration.[1][2][3]
Nomenklatur
Der Name "Assemblin" leitet sich von seiner Funktion ab, das assembly protein (über einen Linker mit dem Gerüstprotein verbundenes Assemblin) zu schneiden. Die Nomenklatur der Assembline, Gerüstproteine und des assembly protein ist in der Literatur uneinheitlich, da diese Proteine nach unterschiedlichen Kriterien benannt wurden und werden. So wurde beispielsweise das HCMV-Assemblin zunächst als "VP24" (virales Protein, 24 kDa) bezeichnet.
Weitere allgemeine Bezeichnungen für die Protease sind "Pr", maturational protease, capsid protease oder einfach protease. Letztgenannter Name kann unter Umständen irreführend sein, da es noch mindestens eine weitere Protease in Herpesviren gibt.[4] Gelegentlich wird auch das Gerüstprotein oder das assembly protein fälschlicherweise als Assemblin bezeichnet.
Funktion
Während des Kapsidzusammenbaus lagern sich Gerüstproteine autokatalytisch aneinander und bilden ein fragiles, sphärisches Prokapsid mit einem definierten Durchmesser.[5][6] Das Proteingerüst besteht aus zwei Genprodukten, dem assembly protein und dem Gerüstprotein, welche im Verhältnis 1:10 vorliegen.[7] Das assembly protein besteht aus zwei Domänen: einer N-terminalen Serinproteasedomäne (Assemblin) und einer C-terminalen Gerüstproteindomäne, welche durch eine Linkerregion miteinander verbunden sind. Das Gerüstprotein entspricht der N-terminalen Domäne des assembly protein. Im Prokapsid dimerisieren die Assemblindomänen durch die räumliche Nähe zueinander. Dadurch wird das Assemblin aktiviert und prozessiert das assembly protein und das Gerüstprotein.[8][9] Es kommt zur Reifung des Kapsids, d. h. durch strukturelle Veränderungen wird aus dem fragilen, sphärischen Prokapsid das stabilere ikosaedrische Kapsid.[10][11] Anschließend wird die virale DNA mithilfe des Terminasekomplexes durch das Portal in die reifen Kapside gepumpt und dabei auf Genomlänge geschnitten.[12][13] Im Zuge dessen wird das Gerüstprotein aus dem Kapsid verdrängt, während das Assemblin im Nukleokapsid verbleibt.
Interessanterweise scheint die Aktivität von freiem Assemblin geringer zu sein, als die des Assemblins als Teil des assembly protein.[14]
Struktur
Die aktive, dimere Struktur von diversen (v. a. humanen) Herpesviren ist bekannt. Die monomere Struktur des um eine Aminosäure C-terminal gekürzten pseudorabies virus-Assemblins ist ebenfalls bekannt.[3]
Das Protein besteht aus einer β-Fass-Struktur, die von zwei β-Faltblättern gebildet wird. Das β-Fass ist umgeben von 6–9 α-Helices. Jedes Monomer trägt eine komplette katalytische Triade, die solvenszugänglich ist. Die Dimergrenzfläche wird von 3 α-Helices je Monomer gebildet und beträgt etwa 1.300 A². Die Ordnung des Dimerisierungsbereichs wird durch die Dimerisierung deutlich erhöht (Unordnung-zu-Ordnung-Mechanismus).[15][16] Dadurch kommt es zur Verschiebung eines Loops (oxyanion-hole loop), welcher in der Dimer-Konformation mit seinem Peptidrückgrat das Oxyanion-Loch bildet. Die katalytische Triade bleibt dabei in Position und Orientierung nahezu unverändert.
In monomeren Assemblinen von Beta- und Gammaherpesviren sind die beiden C-terminalen Helices ungeordnet, während dies bei Alphaherpesviren nicht der Fall ist.[3]
Schnittstellen
Alle Assembline schneiden an mindestens zwei verschiedenen Stellen. Sie schneiden autoproteolytisch an den sogenannten R- und M-sites (release bzw. maturational) des assembly protein und proteolytisch an der M-site des Gerüstproteins. Die R-site befindet sich zwischen der Assemblin- und der Gerüstproteindomäne des assembly protein. Die M-site befindet sich am N-Terminus der Gerüstproteindomäne, wo das Hauptkapsidprotein an das Gerüstprotein gebunden ist.[17][18]
Die Konsensussequenz der R-site ist Y - V/L - K/Q - A | S/N/T, wobei P1' meist Serin ist. Die Konsensussequenz der M-site ist V/L/I - X - A | S. X ist dabei Asn, Gln, Asp oder Glu.[19] Untersuchungen am HCMV-Assemblin ergaben, dass die M-site schneller geschnitten wird, als die R-site.[14]
In assembly proteins einiger Herpesviren (z. B. HCMV) gibt es noch weitere Schnittstellen für das Assemblin, die vermutlich der Regulation dienen.[20][21][22]
Inhibitoren
Assembline werden von den meisten klassischen Serinproteaseinhibitoren kaum gehemmt. Lediglich DFP inhibiert Assembline.[23] Dies geschieht durch eine kovalente Bindung am Serin des aktiven Zentrums. Es wurden außerdem helikale Peptidmimetika gefunden, welche nicht-kovalent an der Dimerisierungsfläche des Monomers binden und eine Dimerisierung und somit Aktivierung des Proteins verhindern.[24]
Einzelnachweise
- ↑ Waxman L, Darke PL: The herpesvirus proteases as targets for antiviral chemotherapy. In: Antivir. Chem. Chemother.. 11, Nr. 1, Januar 2000, S. 1–22. doi:10.1177/095632020001100101. PMID 10693650.
- ↑ Darke PL, Cole JL, Waxman L, Hall DL, Sardana MK, Kuo LC: Active human cytomegalovirus protease is a dimer. In: J. Biol. Chem.. 271, Nr. 13, März 1996, S. 7445–7449. doi:10.1074/jbc.271.13.7445. PMID 8631772.
- ↑ a b c Zühlsdorf M, Werten S, Klupp BG, Palm GJ, Mettenleiter TC, Hinrichs W: Dimerization-Induced Allosteric Changes of the Oxyanion-Hole Loop Activate the Pseudorabies Virus Assemblin pUL26N, a Herpesvirus Serine Protease. In: PLoS Pathog.. 11, Nr. 7, Juli 2015. doi:10.1371/journal.ppat.1005045. PMID 26161660. PMC 4498786 (freier Volltext).
- ↑ Wang S, Wang K, Li J, Zheng C: Herpes simplex virus 1 ubiquitin-specific protease UL36 inhibits beta interferon production by deubiquitinating TRAF3.. In: J. Virol.. 87, Nr. 21, August 2013, S. 11851–11860. doi:10.1128/JVI.01211-13. PMID 23986588. PMC 3807349 (freier Volltext).
- ↑ Newcomb WW, Homa FL, Thomsen DR, Trus BL, Cheng N, Steven A, Booy F, Brown JC: Assembly of the herpes simplex virus procapsid from purified components and identification of small complexes containing the major capsid and scaffolding proteins. In: J. Virol.. 73, Nr. 5, Mai 1999, S. 4239–4250. PMID 10196320. PMC 104203 (freier Volltext).
- ↑ Sheaffer AK, Newcomb WW, Brown JC, Gao M, Weller SK, Tenney DJ: Evidence for controlled incorporation of herpes simplex virus type 1 UL26 protease into capsids. In: J. Virol.. 74, Nr. 15, August 2000, S. 6838–6848. doi:10.1128/JVI.74.15.6838-6848.2000. PMID 10888623. PMC 112201 (freier Volltext).
- ↑ Newcomb WW, Trus BL, Booy FP, Steven AC, Wall JS, Brown JC: Structure of the herpes simplex virus capsid. Molecular composition of the pentons and the triplexes.. In: J. Mol. Biol.. 232, Nr. 2, Juli 1993, S. 499–511. doi:10.1006/jmbi.1993.1406. PMID 8393939.
- ↑ Nomura AM, Marnett AB, Shimba N, Dötsch V, Craik CS: Induced structure of a helical switch as a mechanism to regulate enzymatic activity. In: Nat. Struct. Mol. Biol.. 12, Nr. 11, November 2005, S. 1019–1020. doi:10.1038/nsmb1006. PMID 16244665.
- ↑ Robertson BJ, McCann PJ 3rd, Matusick-Kumar L, Newcomb WW, Brown JC, Colonno RJ, Gao M: Separate functional domains of the herpes simplex virus type 1 protease: evidence for cleavage inside capsids. In: Nat. Struct. Mol. Biol.. 70, Nr. 7, Juli 1996, S. 4317–4328. PMID 8676454. PMC 190364 (freier Volltext).
- ↑ Trus BL, Booy FP, Newcomb WW, Brown JC, Homa FL, Thomsen DR, Steven AC: The herpes simplex virus procapsid: structure, conformational changes upon maturation, and roles of the triplex proteins VP19c and VP23 in assembly. In: J. Mol. Biol.. 263, Nr. 3, November 1996, S. 447–462. doi:10.1016/S0022-2836(96)80018-0. PMID 8918600.
- ↑ Heymann JB, Cheng N, Newcomb WW, Trus BL, Brown JC, Steven AC: Dynamics of herpes simplex virus capsid maturation visualized by time-lapse cryo-electron microscopy. In: Nat. Struct. Biol.. 10, Nr. 5, Mai 2003, S. 334–341. doi:10.1038/nsb922. PMID 12704429.
- ↑ Beard PM, Taus NS, Baines JD: DNA Cleavage and Packaging Proteins Encoded by Genes UL28, UL15, and UL33 of Herpes Simplex Virus Type 1 Form a Complex in Infected Cells. In: J. Virol.. 76, Nr. 10, Mai 2002, S. 4785–4791. doi:10.1128/JVI.76.10.4785-4791.2002. PMID 11967295. PMC 136146 (freier Volltext).
- ↑ White CA, Stow ND, Patel AH, Hughes M, Preston VG: Herpes Simplex Virus Type 1 Portal Protein UL6 Interacts with the Putative Terminase Subunits UL15 and UL28. In: J. Virol.. 77, Nr. 11, Juni 2003, S. 6351–6358. doi:10.1128/JVI.77.11.6351-6358.2003. PMID 12743292. PMC 154995 (freier Volltext).
- ↑ a b Fernandes SM, Brignole EJ, Taori K, Gibson W: Cytomegalovirus Capsid Protease: Biological Substrates Are Cleaved More Efficiently by Full-Length Enzyme (pUL80a) than by the Catalytic Domain (Assemblin). In: J. Virol.. 85, Nr. 7, April 2011, S. 3526–3534. doi:10.1128/JVI.02663-10. PMID 21270147. PMC 3067851 (freier Volltext).
- ↑ Lee GM, Shahian T, Baharuddin A, Gable JE, Craik CS: Enzyme inhibition by allosteric capture of an inactive conformation. In: J. Mol. Biol.. 411, Nr. 5, September 2011, S. 999–1016. doi:10.1016/j.jmb.2011.06.032. PMID 21723875. PMC 3157250 (freier Volltext).
- ↑ Nomura AM, Marnett AB, Shimba N, Dötsch V, Craik CS: Induced structure of a helical switch as a mechanism to regulate enzymatic activity. In: Nat. Struct. Mol. Biol.. 12, Nr. 11, November 2005, S. 1019–1020. doi:10.1038/nsmb1006. PMID 16244665.
- ↑ Preston VG, Rixon FJ, McDougall IM, McGregor M, al Kobaisi MF: Processing of the herpes simplex virus assembly protein ICP35 near its carboxy terminal end requires the product of the whole of the UL26 reading frame. In: Virol.. 186, Nr. 1, Januar 1992, S. 87–98. doi:10.1016/0042-6822(92)90063-U. PMID 1309284.
- ↑ Stevens JT, Mapelli C, Tsao J, Hail M, O'Boyle D 2nd, Weinheimer SP, Diianni CL: In vitro proteolytic activity and active-site identification of the human cytomegalovirus protease. In: Eur. J. Biochem.. 226, Nr. 2, Dezember 1994, S. 361–367. doi:10.1111/j.1432-1033.1994.tb20060.x. PMID 8001553.
- ↑ Gibson W, Welch AR, Hall MRT: Assemblin, a herpes virus serine maturational proteinase and new molecular target for antivirals. In: Perspect. Drug Discov. Des.. 2, Nr. 3, Juli 1995, S. 413–426. doi:10.1007/BF02172034.
- ↑ Holwerda BC, Wittwer AJ, Duffin KL, Smith C, Toth MV, Carr LS, Wiegand RC, Bryant ML: Activity of two-chain recombinant human cytomegalovirus protease. In: J. Biol. Chem.. 269, Nr. 41, Oktober 1994, S. 25911–25915. PMID 7929296.
- ↑ Loveland AN, Chan CK, Brignole EJ, Gibson W: Cleavage of human cytomegalovirus protease pUL80a at internal and cryptic sites is not essential but enhances infectivity. In: J. Virol.. 79, Nr. 20, Oktober 2005, S. 12961–12968. doi:10.1128/JVI.79.20.12961-12968.2005. PMID 16188998. PMC 1235863 (freier Volltext).
- ↑ Pray TR, Nomura AM, Pennington MW, Craik CS: Auto-inactivation by cleavage within the dimer interface of Kaposi’s sarcoma-associated herpesvirus protease. In: J. Mol. Biol.. 289, Nr. 2, Juni 1999, S. 197–203. doi:10.1006/jmbi.1999.2791. PMID 10366498.
- ↑ Burck PJ, Berg DH, Luk TP, Sassmannshausen LM, Wakulchik M, Smith DP, Hsiung HM, Becker GW, Gibson W, Villarreal EC: Human cytomegalovirus maturational proteinase: expression in Escherichia coli, purification, and enzymatic characterization by using peptide substrate mimics of natural cleavage sites. In: J. Virol.. 68, Nr. 5, Mai 1994, S. 2937–2946. PMID 8151764. PMC 236782 (freier Volltext).
- ↑ Shahian T, Lee GM, Lazic A, Arnold LA, Velusamy P, Roels CM, Guy RK, Craik CS: Inhibition of a viral enzyme by a small-molecule dimer disruptor. In: Nat. Chem. Biol.. 5, Nr. 9, September 2009, S. 640–646. doi:10.1038/nchembio.192. PMID 19633659. PMC 2752665 (freier Volltext).
