Vibrio cholerae
| Vibrio cholerae | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|

Vibrio cholerae | ||||||||||||
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| Vibrio cholerae | ||||||||||||
| Pacini 1854 |
Vibrio cholerae (früher Vibrio comma) ist der Erreger der Cholera. Es handelt sich um ein gramnegatives Bakterium aus der Gattung der Vibrionen. Die Zellen sind fakultativ anaerob, sie können mit und ohne Sauerstoff leben. Der Krankheitserreger wurde 1854 von Filippo Pacini als gekrümmtes, kommaförmiges und hochbewegliches Bakterium beschrieben.[1] Im gleichen Jahr beschrieb der Katalane Joaquim Balcells i Pascual den Erreger.[2][3] Robert Koch hat 1884 mit Bernhard Fischer und Georg Gaffky in Indien den Erreger aus dem Darm verstorbener Patienten in Reinkultur angezüchtet.[4]
Die Art umfasst zahlreiche Bakterienstämme.[5] Das Genom des Stammes Vibrio cholerae O1 N16961 (auch als Biovar El Tor bezeichnet)[6] wurde im Jahr 2000 vollständig sequenziert.[7][8] Es verteilt sich auf zwei Segmente, auch Bakterienchromosomen genannt, was für Bakterien ungewöhnlich ist, da die meisten Bakterien nur ein einziges, zirkuläres dsDNA-Molekül besitzen.[9] Nicht alle Bakterienstämme sind pathogen („krankmachend“). Das Bakterium erlangt seine Pathogenität durch Infektion mit bestimmten Bakteriophagen (Viren, die auf Bakterien spezialisiert sind).[10]
Merkmale
Erscheinungsbild
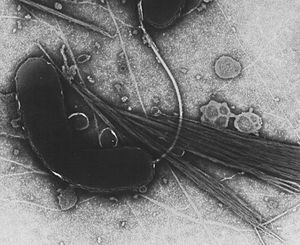
Als typischer Vertreter der Gattung Vibrio zeigt Vibrio cholerae die Zellform eines kommaförmig gekrümmten Stäbchens, das gestalt-bezogen von Robert Koch „Kommabazillus“ genannt[11] wurde. In der Gram-Färbung verhält er sich gramnegativ, wird also durch die verwendeten Farbstoffe rot angefärbt. Verursacht wird dies durch eine dünne Mureinschicht in der Zellwand. Er bewegt sich wie andere Vibrio-Arten mit einer einzelnen Geißel fort. Diese sitzt an nur einem Ende der Bakterienzelle, so dass eine monopolar monotriche Begeißelung vorliegt. Überdauerungsformen wie Endosporen werden nicht gebildet.[12] Die meisten Stämme besitzen keine Kapsel, die der Bakterienzellwand aufgelagert ist, eine Ausnahme bildet die Serogruppe O139, die über eine Kapsel verfügt.[13]
Wachstum und Stoffwechsel
Die Zellen sind fakultativ anaerob, sie können sich also auch vermehren, wenn kein Sauerstoff vorhanden ist. Sie sind Katalase-positiv und Oxidase-positiv, letzteres dient als Unterscheidungsmerkmal zu Vertretern der Enterobacteriaceae.[14] Die Wachstumstemperatur im natürlichen Lebensraum liegt bei 20–30 °C, somit gehört Vibrio cholerae zu den mesophilen (mittlere Temperaturen bevorzugenden) Bakterien.[7] Er kann jedoch auch etwas höhere Temperaturen beim Wachstum tolerieren. Dies nutzt man, falls man ihn im Rahmen einer mikrobiologischen Untersuchung gezielt anzüchtet. Hierbei wird meist eine Temperatur von 35–42 °C zur Kultivierung verwendet.[15] Da V. cholerae im Meerwasser beheimatet ist, ist er halophil („salzliebend“), kann also in Nährmedien mit erhöhter Salzkonzentration kultiviert werden.[16] Außerdem verhält er sich alkalitolerant und kann daher in Nährmedien mit alkalischem pH-Wert wachsen.[17]
V. cholerae betreibt einen chemoorganotrophen und heterotrophen Stoffwechsel, er benutzt organische Verbindungen als Energiequelle und ebenso zum Aufbau zelleigener Stoffe. Sein Stoffwechsel ähnelt dem der Vertreter der Enterobacteriaceae, er kann mehrere Substrate in einer Gärung verwerten.[12] So werden verschiedene Kohlenhydrate (z. B. Glucose, Saccharose und Mannose) fermentativ zu Säuren und anderen Produkten abgebaut. Gas wird dabei nicht gebildet. Außerdem besitzt er die Enzyme Ornithindecarboxylase (ODC) und Lysindecarboxylase (LDC), die die Abspaltung von Kohlenstoffdioxid (CO2) bei den Aminosäuren Ornithin und Lysin ermöglichen.[15] Daher kann auch eine „Bunte Reihe“, die zur Unterscheidung der Enterobacteriaceae verwendet wird, für die Bestimmung von V. cholerae eingesetzt werden.
Genetik

Das Genom des Stammes Vibrio cholerae O1 N16961 (auch als Biovar El Tor bezeichnet) wurde im Jahr 2000 vollständig sequenziert. Der für die Untersuchung verwendete Bakterienstamm wurde aus einer Stuhlprobe eines Patienten einer Choleraepidemie in Bangladesch 1971 isoliert. Das Genom weist eine Größe von 4033 Kilobasenpaaren (kb) auf,[7] was in etwa mit der Genomgröße von Escherichia coli vergleichbar ist. Es sind 3887 Proteine annotiert.[8] Die Ergebnisse der Sequenzierungen des El-Tor-Stammes und weiterer Stämme der Art zeigen einen GC-Gehalt (den Anteil der Nukleinbasen Guanin und Cytosin) in der Bakterien-DNA von 47–48 Mol-Prozent.[18] Dies ist vergleichbar mit dem GC-Gehalt in der DNA von E. coli und anderer Enterobacteriaceae, die wie Vibrio zur Klasse der Gammaproteobacteria gehören.[19]
Das Genom von V. cholerae verteilt sich auf zwei zirkuläre Segmente, oft auch Bakterienchromosome genannt. Dies ist für Bakterien ungewöhnlich, da die meisten Bakterien nur ein einziges kovalent geschlossenes, ringförmiges dsDNA-Molekül besitzen.[9] Chromosom 1 von V. cholerae umfasst 2961 kb, während Chromosom 2 mit 1072 kb kleiner ausfällt. Die meisten Gene für wichtige Zellfunktionen, wie Replikation, Transkription, Translation der DNA sowie Synthese der Bakterienzellwand, aber auch die Virulenzfaktoren befinden sich auf dem großen Chromosom. Das kleinere Chromosom umfasst Gene, die nicht typisch für das Genom der Gammaproteobacteria sind. Vielmehr handelt es sich um Gene, die typischerweise auf einem Plasmid lokalisiert sind. So findet sich hier beispielsweise ein Integron (als Integron Island bezeichnet). Dieser Genabschnitt dient dazu, Gene aus einem Chromosom oder einem Plasmid einzufangen, was für die Pathogenität von Bedeutung ist. Da derartige Integrons sonst nur auf einem Plasmid enthalten sind, führte ihre Entdeckung auf dem Bakterienchromosom 2 zu der Annahme, dass sein Ursprung ein „Megaplasmid“ ist, das von der Urform einer Vibrio-Art aufgenommen wurde.[9]
Nachweise

Die in der Lebensmittelmikrobiologie eingesetzten Untersuchungsmethoden für Vibrio cholerae und andere Vibrio-Arten sind durch die ISO 21872[20] und in den USA durch das Bacteriological Analytical Manual (bakteriologisch-analytisches Handbuch, Abkürzung BAM) der Food and Drug Administration (FDA) – der US-amerikanischen Behörde für Lebensmittel- und Arzneimittelsicherheit – vorgeschrieben.[15]
Bei Proben wie Wasser oder Lebensmitteln erfolgt zunächst eine Anreicherung von V. cholerae in alkalischem Peptonwasser. Neben Pepton (einem Gemisch aus Peptiden und Aminosäuren) weist diese Nährbouillon eine hohe Konzentration an Natriumchlorid sowie einen alkalischen pH-Wert von pH 8,5 auf, durch diese beiden Parameter wird das Wachstum zahlreicher anderer Bakterien gehemmt.[21] Falls die Kultivierung in diesem Medium bei 42 °C erfolgt, ist die Anreicherung noch selektiver,[15] da durch die hohe Temperatur andere mesophile Bakterien im Wachstum gehemmt werden. Zur Isolierung von V. cholerae wird nach der Anreicherung ein geringes Volumen der Nährbouillon auf TCBS-Agar ausplattiert, der als Vibrio-Selektivmedium verwendet wird.
Bei klinischen Proben, wie z. B. Stuhlproben oder Erbrochenem, können diese mit einem Tupfer auf dem Selektivmedium verteilt werden. Allerdings empfiehlt sich auch hier eine Selektionierung mit einem alkalischen Medium wie dem alkalischen Peptonwasser.[17] Neben dem Selektivmedium TCBS-Agar sollte auch ein wenig selektiv wirkendes Nährmedium beimpft werden.[22] Es wird auch der direkte Nachweis der beweglichen Vibrionen in klinischen Proben mit Hilfe der Dunkelfeldmikroskopie oder der Phasenkontrastmikroskopie durchgeführt,[16] wobei auf diese Weise aber keine sichere Identifizierung der Art möglich ist.
TCBS-Agar steht für Thiosulfate Citrate Bile Sucrose Agar und verweist auf die wichtigsten Komponenten des Nährmediums: Hohe Konzentrationen an Natriumthiosulfat und Natriumcitrat hemmen weitgehend das Wachstum von gramnegativen Enterobacteriaceae, während Ochsengalle (im Englischen bile) das Wachstum der grampositiven Begleitflora, vor allem der Enterokokken verhindert. Saccharose (im Englischen sucrose) ist das einzige Kohlenhydrat im TCBS-Agar und schränkt das Wachstum für Bakterien ein, die Saccharose nicht verwerten können. Zusammen mit den enthaltenen pH-Indikatoren kann der Abbau des Kohlenhydrats durch Vibrio-Arten über die Säurebildung sichtbar gemacht werden. Unbeimpft weist das Medium einen alkalischen pH-Wert (pH 8,6) auf, durch den ebenfalls eine Wachstumshemmung anderer Bakterien erfolgt.[23]
Auf TCBS-Agar gewachsene Kolonien müssen zur Differenzierung der verschiedenen Vibrio-Arten noch weiter untersucht werden. Biochemische Tests zur Identifizierung beinhalten, wie bereits beschrieben, den Katalase- und Oxidase-Test, sowie typische Tests aus einer „Bunten Reihe“, wobei unter anderem auf die Verwertbarkeit verschiedener Kohlenhydrate und anderer Substrate untersucht wird. Ein darauf basierendes Schnellbestimmungssystem im Miniaturformat (Analytical Profile Index) zur Bestimmung von Bakterien aus den Familien Enterobacteriaceae und Vibrionaceae ist kommerziell verfügbar.[24]
Die Zuordnung zu den Serotypen kann mittels eines Agglutinationstests erfolgen. Dabei wird ein polyvalentes O-spezifisches Antiserum eingesetzt, das mit Probematerial, welches O-Antigene von V. cholerae enthält, zur Agglutination führt.[17] Dieses Verfahren kann als so genannter Latextest (Latex-Agglutinationstest) durchgeführt werden, bei dem die Antigen-Antikörper-Reaktion mit Hilfe von Latexpartikeln sichtbar gemacht wird. Der Nachweis von V. cholerae kann auch direkt mit Hilfe des ELISA-Verfahrens (quantitativer Nachweis der Antigene) durchgeführt werden. Dabei werden sowohl die toxinbildenden Stämme der Serogruppen O1 und O139 wie auch nicht toxinbildende Stämme erfasst. Als Probe kann ein rektaler Abstrich eines Patienten verwendet werden, genauso lassen sich auch Proben aus der Umwelt – beispielsweise Wasserproben – untersuchen.[25]
Neben dem Nachweis des Bakteriums erfolgt auch der Nachweis des durch den Erreger gebildeten Choleratoxins. Dies geschieht mit Hilfe des bereits genannten Latex-Agglutinationstests. Die benötigten Antikörper erhält man aus dem Blutserum von Kaninchen, die mit dem gereinigten Choleratoxin immunisiert wurden. Derartige Testsysteme werden als RPLA (reversed passive latex agglutination; übersetzt „umgekehrte, passive Latex-Agglutination“) bezeichnet und sind als gleichzeitige Nachweismethode des Choleratoxins und des hitzelabilen Enterotoxins LT der Enterotoxischen Escherichia coli (ETEC) kommerziell verfügbar.[26] Der Test wird mit Mikrotiterplatten durchgeführt, als Probe werden verdünnte Flüssigkulturen eingesetzt, zu denen die Latexsuspension hinzugegeben wird.[27] Die Nachweisgrenze liegt bei 1–2 ng/ml, die Sensitivität und die Spezifität des RPLA-Tests sind vergleichbar mit der ELISA-Methode.[28] Ähnlich spezifisch ist der Nachweis bestimmter Teile des bakteriellen Genoms mit Hilfe des PCR-Verfahrens (Polymerase-Kettenreaktion). Hierbei ist der Teil des Genoms, in dem die Toxinbildung codiert ist, Ziel der Untersuchung. Der Nachweis erfolgt mit Hilfe des Multiplex-PCR Verfahrens, dabei ist auch die gleichzeitige Unterscheidung von anderen Enterotoxinen, die Gastroenteritis verursachen, möglich.[29]
Vorkommen

Vibrio cholerae ist ein aquatisches Bakterium, er kommt also im Wasser vor, sowohl im Süßwasser als auch im Meerwasser, wobei hier vor allem die Brack- und Küstengewässer von Bedeutung sind. Durch das Wasser erfolgt auch die Übertragung auf den Menschen. Insbesondere nicht oder unzureichend aufbereitetes Trinkwasser ist der Grund für die Übertragung, sowie Lebensmittel, die mit kontaminiertem Wasser in Berührung gekommen sind.[30] Auch in Deutschland wurde Vibrio cholerae vereinzelt in stehenden Gewässern – v. a. bei Wassertemperaturen > 20 °C – nachgewiesen. Die gefundenen Vibrionen waren keine Choleratoxinbildner.[31]
Auch pflanzliche Lebensmittel können mit Vibrio cholerae kontaminiert werden, wenn Fäkalien mit dem Erreger als Düngemittel auf Feldern aufgebracht oder wenn die Lebensmittel zur Frischhaltung mit kontaminiertem Wasser benetzt werden. Häufiger jedoch ist das Bakterium in tierischen Lebensmitteln zu finden, die dem Meer entstammen.[32] Bei an Cholera erkrankten Patienten ist der Erreger im Stuhl, im Erbrochenen und im Duodenalsaft nachweisbar.[33] Nach überstandener Erkrankung ist V. cholerae noch einige Wochen lang im Stuhl nachweisbar, Dauerausscheider sind aber selten.[16]
Systematik
Äußere Systematik
Vibrio cholerae ist ein typischer Vertreter der Gattung Vibrio, die umgangssprachlich auch als Vibrionen bezeichnet werden. Neben dem Choleraerreger sind auch die Arten V. parahaemolyticus, Vibrio vulnificus und Vibrio alginolyticus von medizinischer Bedeutung.[16] Neben den Vibrionen gibt es noch weitere Gattungen, die der Familie der Vibrionaceae angehören, während diese die einzige Familie in der Ordnung Vibrionales darstellt.
Innere Systematik
Die Spezies umfasst zahlreiche Stämme.[5] Unterscheidet man Vibrio cholerae nach den O-Antigenen, lassen sich über 150 Serotypen erfassen.[15] Als Erreger der Cholera werden die Serogruppen V. cholerae O1[34] und O139[35] angesehen, wobei die Serogruppe O1 für die meisten Ausbrüche von Cholera verantwortlich ist. Zu dieser Gruppe zählt man den klassischen Biotyp und den Biotyp El Tor[16][6] (siehe unten). Beide Biotypen weisen jeweils zwei eigene Serotypen auf, die als Inaba[36] und Ogawa[37] bezeichnet werden.[38]
Für die Choleraepidemien im 19. und 20. Jahrhundert wird der klassische Biotyp als Erreger angesehen, der einen deutlich schwerwiegenderen Krankheitsverlauf hervorruft. Seit den 1970er Jahren werden überwiegend Infektionen durch den Biotyp (auch als Biovar bezeichnet) El Tor beobachtet, die meist weniger schwerwiegend verlaufen. Lediglich in Bangladesch werden auch seit 1982 noch Ausbrüche durch den klassischen Biotyp dokumentiert, daher wird die südliche Küstenregion von Bangladesch als Habitat des klassischen Biotyps angesehen.[38] Aus jener Zeit stammt der Begriff der so genannten NAG-Vibrionen (nicht agglutinierende Vibrionen), also Stämme, die mit einem O-spezifischen Antiserum nicht agglutinieren. Tatsächlich bezieht sich die Nichtagglutination aber nur auf das Antigen O1, das zu der Zeit das einzig bekannte O-Antigen war. Diese Non-O1-Stämme von Vibrio cholerae verursachen ebenfalls Gastroenteritis, allerdings ging man damals davon aus, dass nur die Serogruppe O1 an Epidemien beteiligt war.[17]
Dies änderte sich 1992, als mehrere Cholera-Ausbrüche im Süden und Osten Indiens sowie im südlichen Bangladesch registriert wurden, die sich in den Südosten Asiens verbreiteten. Als Erreger wurden Stämme der Serogruppe O139 (V. cholerae O139 Synonym Bengal) identifiziert, ein bis dahin unbekanntes Serovar. Diese Stämme zeigen keine Agglutination mit polyklonalen oder monoklonalen Antikörpern, die gegen das V.-cholerae-O1-Antigen gerichtet sind. Studien aus dem Epidemiegebiet führten außerdem zu der Annahme, dass eine gegen V. cholerae O1 El Tor erworbene Immunität nicht vor einer Ansteckung mit V. cholerae O139 schützt. Diese Bengal-Stämme verfügen im Gegensatz zu der Serogruppe O1 über eine Kapsel, außerdem zeigen Teile des für das O1-Antigen verantwortlichen Genkomplexes eine veränderte Struktur oder fehlen ganz.[13] Neben den Serogruppen V. cholerae O1 und O139 gibt es auch noch so genannte Non-O1- und Non-O139-Stämme, die ebenfalls Erkrankungen verursachen, die aber nicht als Ausbruch von Cholera beschrieben werden.[16]
Etymologie
Otto Friedrich Müller, ein dänischer Naturforscher, entwickelte 1786 erste Versuche zur Klassifizierung und Beschreibung von Mikroorganismen und prägte für bewegliche Mikroorganismen den Begriff „Zittertierchen“ oder Vibrionen (vibrare aus dem Lateinischen bedeutet „sich schnell hin- und herbewegend“, „vibrierend“). Der Artname verweist auf die durch das Bakterium hervorgerufene Krankheit Cholera, entstammt dem Altgriechischen und bedeutet „Fluss der gelben Galle“.[17] Der Biotyp El Tor ist benannt nach der Kleinstadt At-Tur (arabisch الطور, latinisiert auch Al-Tur, El Tor), Hauptstadt des Süd-Sinai ![]() . Dort isolierte der deutsche Bakteriologe Felix Gotschlich 1905 den Biotyp in einer Quarantäne-Station. Bei den Patienten der Quarantäne-Station handelte es sich um Pilger, die aus Mekka zurückgekehrt waren.[33]
. Dort isolierte der deutsche Bakteriologe Felix Gotschlich 1905 den Biotyp in einer Quarantäne-Station. Bei den Patienten der Quarantäne-Station handelte es sich um Pilger, die aus Mekka zurückgekehrt waren.[33]
Medizinische Bedeutung
Pathogenität
Die Pathogenität von Vibrio cholerae beruht zum einen auf der Freisetzung eines Exotoxins, das als Enterotoxin auf den Darm wirkt. Dieses sogenannte Choleratoxin (CTX) ist für die Symptome der Infektionskrankheit Cholera verantwortlich. Die Bildung des Choleratoxins ist im Genom des Bakteriums codiert, allerdings gilt das nur für die pathogenen Stämme, wie die Serogruppen V. cholerae O1 und O139.[39]
Im Bakterienchromosom finden sich auf einer sogenannten Pathogenitätsinsel die Gene, die verschiedene Virulenzfaktoren codieren, bei V. cholerae wird diese Pathogenitätsinsel als VPI (V. cholerae Pathogenicity Island) bezeichnet. Auf der VPI finden sich das tcpA-Gen für den TCP-Faktor (
), dadurch bildet das Bakterium spezielle Pili (Typ IV Pili) aus.[10] Diese sind den Geißeln ähnelnde Oberflächenstrukturen aus Proteinen, die jedoch anders als diese nicht zur aktiven Bewegung dienen.[40] Diese filamentösen Zelloberflächenstrukturen wirken als Adhäsine und ermöglichen somit das Anheften der Bakterienzelle an der Oberfläche der Mikrovilli der Darmzellen. Somit sind sie ein weiterer Virulenzfaktor.[39][10] Es wird vermutet, dass die Gene der Pathogenitätsinsel von einem Bakteriophagen (in der Kurzform auch Phage genannt) stammen. In einer Untersuchung aus dem Jahr 1999 wurde gezeigt, dass die tcp Gene der VPI identisch sind mit denen eines Phagen, der folglich als VPIΦ („
“ VPIphi,
) bezeichnet wird.[10] VPIΦ ist demzufolge verantwortlich für den horizontalen Gentransfer zwischen V. cholerae Stämmen. Weiterhin wurde die TcpA-Untereinheit des Typ IV Pilus als Kapsidprotein des Bakteriophagen VPIΦ erkannt.[10]

Der Typ IV Pilus dient außerdem als Rezeptor für einen weiteren Bakteriophagen, der als CTXΦ (CTXphi, Choleratoxin-Phage, Spezies Vibrio-Virus CTXphi in der Familie Inoviridae, Gattung Affertcholeramvirus) bezeichnet wird.[10] In seinem Genom finden sich die ctxAB-Gene, die für die Untereinheiten A und B des Choleratoxins codieren.[39] CTXΦ ist ein temperenter Phage, der in das Bakterienchromosom integriert. Die DNA des Phagen wird in das Chromosom des Bakteriums eingebaut – dies wird dann als Prophage bezeichnet – und bei jeder folgenden Zellteilung werden die Gene des Prophagen und die des Bakteriums gemeinsam verdoppelt und weitergegeben. Dadurch bildet das Bakterium den Accessory Colony Factor (ACF) und es kommt zur Ausschüttung des Choleratoxins. Üblicherweise bleibt es bei diesem sogenannten lysogenen Zyklus, es kommt also nicht zu einer Aktivierung des lytischen Zyklus, die Phagen lysieren die Zelle nicht, und so verbleibt CTXΦ als Prophage in der Zelle und vermehrt sich mit.[41]
Nur wenn ein V.-cholerae-Stamm über beide Virulenzfaktoren verfügt, ist er als pathogener Stamm anzusehen. In einer genetischen Untersuchung von 300 nichtpathogenen Stämmen konnte lediglich bei 15 Stämmen eine VPI mit einem tcpA-Gen erkannt werden, davon ließ sich bei neun Stämmen auch CTXΦ als Prophage nachweisen. Keiner der untersuchten Stämme wies nur die ctxAB-Gene auf, was bestätigt, dass der Phage CTXΦ nur in das Bakterienchromosom integriert, wenn bereits das tcpA-Gen in der VPI vorhanden ist. Die untersuchten Stämme, die VPI und CTXΦ als Prophagen aufweisen, gehören anderen Serogruppen als O1 und O139 an und sind nicht an Choleraepidemien beteiligt. Dies wird durch Abweichungen in der DNA-Sequenz der VPI bzw. der Prophagen begründet. Daraus wird geschlussfolgert, dass sich diese nichtepidemischen Stämme aus nichtpathogenen V. cholerae entwickelt haben, die durch horizontalen Gentransfer VPI und CTXΦ in ihr Genom aufgenommen haben.[39]
Die Herkunft der ctxAB-Gene durch den Phagen CTXΦ wurde bereits bewiesen, so konnte eine Übertragung der Gene zwischen V.-cholerae-Stämmen (also innerhalb einer Spezies) mit Hilfe des CTXΦ gezeigt werden. Dieser – durch einen Phagen verursachte – horizontale Gentransfer wird dann als Transduktion bezeichnet. Auch die Übertragung zwischen Spezies ist möglich, beispielsweise von V. cholerae auf Vibrio mimicus. Weiterhin weist die VPI von V. mimicus bei einzelnen Genen eine identische DNA-Sequenz mit der von V. cholerae O1 N16961 auf, was als horizontaler Gentransfer durch den Phagen VPIΦ erklärt wird.[42]
Die Herkunft der Gene in der Pathogenitätsinsel ist jedoch noch nicht abschließend geklärt. Normalerweise kann man durch bestimmte Reize einen Prophagen wieder aktivieren, in der Form, dass seine Gene „abgelesen“ werden. Dies führt bei temperenten Phagen dazu, dass aus dem lysogenen Zyklus ein lytischer Zyklus wird, bei dem anschließend die Phagen freigesetzt werden.[43] Behandelt man die Prophagen von V. cholerae mit Mitomycin C oder mit UV-Strahlung, so erfolgt eine Induktion des lytischen Zyklus. In einer darauf basierenden Untersuchung aus dem Jahr 2003 ließ sich sowohl bei V.-cholerae-O139-Stämmen wie auch beim Biovar El Tor die Produktion von extrazellulären CTXΦ nachweisen. Dies gelang jedoch nicht für den Phagen VPIΦ. Damit ist noch nicht endgültig bewiesen, ob der horizontale Gentransfer der VPI tatsächlich auf einem Phagen beruht oder auf einem anderen Mechanismus.[44]
Unabhängig von der vorliegenden Serogruppe wird V. cholerae durch die Biostoffverordnung in Verbindung mit der TRBA (Technische Regeln für Biologische Arbeitsstoffe) 466 der Risikogruppe 2 zugeordnet.[45]
Infektionsquellen
Der Infektionsweg von Vibrio cholerae erfolgt immer oral, in den meisten Fällen geschieht die Aufnahme über kontaminiertes Trinkwasser, zum Teil auch über Lebensmittel. Selten erfolgt die direkte fäkal-orale Übertragung von Mensch zu Mensch.[16] Auch auf Planktonbestandteilen und Süßwasseralgen[30] lässt sich der Erreger nachweisen. Durch sie werden dann Fische und Meeresfrüchte kontaminiert, die roh oder unzureichend gegart die Infektion des Menschen ermöglichen, der diese Lebensmittel zu sich nimmt. Das Bakterium kann bei genügend Feuchtigkeit mehrere Wochen überleben.[16] Allerdings haben Studien an Freiwilligen gezeigt, dass eine recht große Menge an V. cholerae aufgenommen werden muss, um eine Infektion mit Cholera hervorzurufen. Die Magensäure reduziert die Anzahl der Erreger, so dass das Inokulum etwa 108 bis 109 Bakterien enthalten muss. Falls V. cholerae mit Nahrungsmitteln aufgenommen wird, ist die Infektionsdosis weitaus geringer,[30] in diesem Fall genügt die Aufnahme von 104 bis 106 Bakterien. Bei Hypoacidität – wenn also beispielsweise durch Medikamentenwirkung weniger Magensäure vorhanden ist – beträgt die Infektionsdosis sogar nur 103 bis 104 Erreger. Wenn sie in den Dünndarm gelangen, können sie sich dort wegen des vorherrschenden alkalischen Milieus wieder vermehren.[33]
Infektionskrankheiten
Vibrio cholerae heftet sich an die Epithelzellen des Dünndarms, vermehrt sich dort und setzt ein Exotoxin frei, das als Enterotoxin wirkt. Die Folge ist eine Gastroenteritis mit sehr starkem Durchfall („Reiswasserstuhl“), der unbehandelt zur Exsikkose (Austrocknung des Körpers) mit Elektrolytverlust und somit zum Tode führen kann. Infektionen durch den Biotyp El Tor verlaufen meistens leichter. Darüber hinaus kommen milde Verläufe ohne typische Symptome vor, bei denen trotzdem der Erreger nachweisbar ist.[16]
Einige der Non-O1- und Non-O139-Stämme führen vereinzelt zu Gastroenteritis, die den Symptomen der Cholera entfernt ähnelt. Untersuchungen solcher Stämme haben gezeigt, dass sich der von ihnen produzierte Permeabilitätsfaktor immunologisch vom Choleratoxin unterscheidet. Andere der Non-O1- und Non-O139-Stämme produzieren ein hitzestabiles Exotoxin, das zu einer septischen Infektion bei Patienten mit bestimmten Vorerkrankungen geführt hat. Der Hauptunterschied liegt darin, dass all diese Stämme kein Choleratoxin freisetzen.[46]
In Deutschland ist der direkte oder indirekte Nachweis von Vibrio cholerae namentlich meldepflichtig nach § 7 des Infektionsschutzgesetzes. nach § 6 IfSG sind bereits der Krankheitsverdacht sowie die Erkrankung an Cholera und der Tod namentlich zu melden, nach § 42 gilt ein Tätigkeits- und Beschäftigungsverbot für die betroffene Person in bestimmten Lebensmittelbetrieben.[47]
In Österreich ist nach § 1 Abs. 1 Epidemiegesetz 1950 Cholera meldepflichtig bezüglich Verdachts-, Erkrankungs- und Todesfällen.
In der Schweiz ist der positive laboranalytische Befund zu Vibrio cholerae meldepflichtig und zwar nach dem Epidemiengesetz (EpG) in Verbindung mit der Epidemienverordnung und Anhang 3 der Verordnung des EDI über die Meldung von Beobachtungen übertragbarer Krankheiten des Menschen.
Therapie und Vorbeugung
Eine Therapie mit Antibiotika, wie beispielsweise mit Ciprofloxacin,[16] kann den Verlauf der Krankheit verkürzen, ist alleine jedoch nicht ausreichend. Gleichzeitig muss für Flüssigkeits- und Elektrolytersatz gesorgt werden, da deren Verluste im Verlauf der Cholera unbehandelt zu einer Mortalitätsrate von 60 % führen.[33] Es ist auch eine Impfung gegen Cholera möglich, bei der ein Totimpfstoff (inaktivierte Zellen von V. cholerae) verwendet wird, der oral als Schluckimpfung verabreicht wird.[48]
Lebensmittelmikrobiologische Bedeutung
Für Trinkwasser als wichtiges Lebensmittel gilt in Deutschland die Trinkwasserverordnung. Hiernach dürfen im Trinkwasser Krankheitserreger „nicht in Konzentrationen enthalten sein, die eine Schädigung der menschlichen Gesundheit besorgen lassen“ (§ 5 Absatz 1 TrinkwV 2001).[49] Die Wasseraufbereitung bei der Produktion von Trinkwasser schützt in Europa vor dem Befall mit Vibrio cholerae. In Ländern oder Regionen mit niedrigem Hygienestandard – verursacht insbesondere durch unzureichende Abwasserentsorgung – ist das Vorkommen des Choleraerregers in Trinkwasser oder Wasser, das für den menschlichen Gebrauch verwendet wird, eine Gefahr. Das Robert Koch-Institut empfiehlt bei Reisen in diese Regionen nur abgekochtes Wasser oder fertig abgepacktes Mineralwasser zum Trinken, Zähneputzen oder zum Geschirrspülen zu verwenden. Eine Impfung für Reisende wird vom Robert Koch-Institut (mit Verweis auf die Weltgesundheitsorganisation WHO) nicht generell empfohlen.[16]

Eine Übertragung von V. cholerae ist auch über Fische und Meeresfrüchte möglich. Die Deutsche Gesellschaft für Hygiene und Mikrobiologie (DGHM e. V.) empfiehlt deshalb bei Seefisch aus wärmeren Regionen die lebensmittelmikrobiologische Untersuchung auf Vibrionen. Falls pathogene Arten nachgewiesen werden, sind weitere Untersuchungen zum Toxinbildungsvermögen notwendig.[50] Der Erreger findet sich auch auf Planktonbestandteilen und Süßwasseralgen[30] und kann somit zu einer Kontamination von Fischen und Meeresfrüchten führen. Daher sollte man zur Prophylaxe auf rohe oder unzureichend gegarte Lebensmittel dieser Art verzichten. Dies gilt insbesondere für Gebiete, in denen mit dem Auftreten des Erregers zu rechnen ist. Ebenso sollte man auf rohe Salate verzichten, da sie mit kontaminiertem Wasser gewaschen sein könnten.[16] Die Regel „Boil it, cook it, peel it, or forget it!“[51] („Koch’ es, gar’ es, schäl’ es oder vergiss es!“) gilt als Richtlinie zur Cholera-Prophylaxe bei Reisen in gefährdete Gebiete.
Eine Untersuchung aus dem Jahre 1995 zeigte, dass zahlreiche Lebensmittel, wenn sie absichtlich mit dem Erreger kontaminiert werden, das Wachstum oder zumindest das Überdauern der Bakterien ermöglichen, in einigen Fällen bis zu drei Monate. Bei den untersuchten Lebensmitteln handelte es sich unter anderem um Joghurt, Milch, Marmelade, Salat, Pasta und Würstchen. Dabei blieben die Vibrionen insbesondere bei einem neutralen oder nur leicht sauren pH-Wert des betreffenden Nahrungsmittels überlebensfähig.[32]
Quellen
Literatur
- Michael T. Madigan, John M. Martinko, Jack Parker: Brock Mikrobiologie. Deutsche Übersetzung herausgegeben von Werner Goebel, 1. Auflage. Spektrum Akademischer Verlag GmbH, Heidelberg/Berlin 2000, ISBN 978-3-8274-0566-1.
- Herbert Hof, Rüdiger Dörries: Duale Reihe: Medizinische Mikrobiologie. 3. Auflage. Thieme Verlag, Stuttgart 2005, ISBN 978-3-13-125313-2.
- Helmut Hahn, Stefan H. E. Kaufmann, Thomas F. Schulz, Sebastian Suerbaum (Hrsg.): Medizinische Mikrobiologie und Infektiologie. 6. Auflage. Springer Verlag, Heidelberg 2009, ISBN 978-3-540-46359-7.
- I. Kaye Wachsmuth, Paul A. Blake, Orjan Olsvik (Hrsg.): Vibrio cholerae and cholera: Molecular to global perspectives. 1. Auflage. ASM Press, Washington 1994, ISBN 1-55581-067-5.
Einzelnachweise
- ↑ M. Bentivoglio, P. Pacini: Filippo Pacini: a determined observer. In: Brain research bulletin. Band 38, Nummer 2, 1995, S. 161–165, doi:10.1016/0361-9230(95)00083-Q. PMID 7583342.
- ↑ Real Academia de la Historia (Hrsg.): Joaquín Balcells y Pasqual, 2018, Archiv-Link (spanisch)
- ↑ Col·legi Oficial de Metges de Barcelona (Hrsg.): Joaquim Balcells i Pascual, 2015, Archiv-Link (katalanisch)
- ↑ Norman Howard-Jones: Robert Koch and the cholera vibrio: a centenary. In: British medical journal (Clinical research ed.). Band 288, Nummer 6414, Februar 1984, S. 379–381, PMID 6419937. PMC 1444283 (freier Volltext).
- ↑ a b Taxonomy Browser Vibrio cholerae. In: Webseite des National Center for Biotechnology Information (NCBI). Abgerufen am 8. März 2013.
- ↑ a b NCBI: Vibrio cholerae O1 biovar El Tor (no rank)
- ↑ a b c Vibrio cholerae O1 bv El Tor, N16961. In: Webseite Genomes Online Database (GOLD). Abgerufen am 8. März 2013.
- ↑ a b Vibrio cholerae El Tor N16961 Genome Page. (Nicht mehr online verfügbar.) In: Webseite J. Craig Venter Institute (JCVI). Archiviert vom Original am 23. Februar 2014; abgerufen am 8. März 2013.
- ↑ a b c John F. Heidelberg, Jonathan A. Eisen u. a.: DNA sequence of both chromosomes of the cholera pathogen Vibrio cholerae. In: Nature. Band 406, Nummer 6795, August 2000, S. 477–483, doi:10.1038/35020000. PMID 10952301.
- ↑ a b c d e f David K. R. Karaolis, Sita Somara, David R. Maneval, Judith A. Johnson, James B. Kaper: A bacteriophage encoding a pathogenicity island, a type-IV pilus and a phage receptor in cholera bacteria. In: Nature. Band 399, Nummer 6734, Mai 1999, S. 375–379, doi:10.1038/20715. PMID 10360577.
- ↑ Gundolf Keil: Robert Koch (1843–1910). Ein Essai. In: Medizinhistorische Mitteilungen. Zeitschrift für Wissenschaftsgeschichte und Fachprosaforschung. Band 36/37, 2017/2018 (2021), S. 73–109, hier: S. 74.
- ↑ a b Hans G. Schlegel, Christiane Zaborosch: Allgemeine Mikrobiologie. 7. Auflage. Thieme Verlag, Stuttgart/New York 1992, ISBN 3-13-444607-3, S. 24, 65, 117.
- ↑ a b J. A. Johnson, C. A. Salles u. a.: Vibrio cholerae O139 synonym bengal is closely related to Vibrio cholerae El Tor but has important differences. In: Infection and immunity. Band 62, Nummer 5, Mai 1994, S. 2108–2110, PMID 8168977. PMC 186475 (freier Volltext).
- ↑ Michael T. Madigan, John M. Martinko, Jack Parker: Brock Mikrobiologie. Deutsche Übersetzung herausgegeben von Werner Goebel, 1. Auflage. Spektrum Akademischer Verlag GmbH, Heidelberg/Berlin 2000, ISBN 978-3-8274-0566-1, S. 536–538.
- ↑ a b c d e Charles A. Kaysner, Angelo DePaola, Jr.: Bacteriological Analytical Manual, chapter 9: Vibrio. In: Website der Food and Drug Administration (FDA). Abgerufen am 18. März 2013.
- ↑ a b c d e f g h i j k l Steckbriefe seltener und importierter Infektionskrankheiten. (PDF) (Nicht mehr online verfügbar.) In: Website des Robert Koch-Instituts (RKI). 15. September 2011, S. 61–62, archiviert vom Original am 30. Dezember 2013; abgerufen am 6. November 2013.
- ↑ a b c d e Herbert Hof, Rüdiger Dörries: Duale Reihe: Medizinische Mikrobiologie. 3. Auflage. Thieme Verlag, Stuttgart 2005, ISBN 978-3-13-125313-2, S. 400–404.
- ↑ Vibrio cholerae. In: Webseite Genome des National Center for Biotechnology Information (NCBI). Abgerufen am 1. November 2013.
- ↑ Michael T. Madigan, John M. Martinko, Jack Parker: Brock Mikrobiologie. Deutsche Übersetzung herausgegeben von Werner Goebel, 1. Auflage. Spektrum Akademischer Verlag GmbH, Heidelberg/Berlin 2000, ISBN 978-3-8274-0566-1, S. 534.
- ↑ Rapid testing solutions for the detection of Vibrio cholerae. (Nicht mehr online verfügbar.) In: Webseite Merck Millipore. Archiviert vom Original am 3. November 2013; abgerufen am 2. November 2013.
- ↑ Datenblatt ENDO-Agar für die Mikrobiologie bei Merck, abgerufen am 2. November 2013.
- ↑ F. Burkhardt: Die bakteriologische Diagnose der Vibrio EI Tor-Infektion. In: Zentralblatt für Bakteriologie, Parasitenkunde, Infektionskrankheiten und Hygiene. 1. Abt. Medizinisch-hygienische Bakteriologie, Virusforschung und Parasitologie. Originale. Band 212, Nummer 1, Dezember 1969, S. 177–189, PMID 4195371.
- ↑ Datenblatt TCBS-Agar (Vibrio-Selektivagar) bei Merck, abgerufen am 2. November 2013.
- ↑ ID 32 biochemische Identifizierung (rapid ID 32 E); Vibrionaceae, Enterobacteriaceae. (Nicht mehr online verfügbar.) In: Webseite der bioMérieux Deutschland GmbH. Archiviert vom Original am 5. Januar 2014; abgerufen am 4. März 2013.
- ↑ U. Tuteja, S. Kumar u. a.: Simultaneous direct detection of toxigenic and non-toxigenic Vibrio cholerae from rectal swabs and environmental samples by sandwich ELISA. In: Journal of medical microbiology. Band 56, Nummer 10, Oktober 2007, S. 1340–1345, doi:10.1099/jmm.0.47166-0. PMID 17893171.
- ↑ D. L. Evers, J. He, J. T. Mason, T. J. O'Leary: The liposome PCR assay is more sensitive than the Vibrio cholerae enterotoxin and Escherichia coli heat-labile enterotoxin reversed passive latex agglutination test at detecting cholera toxin in feces and water. In: Journal of clinical microbiology. Band 48, Nummer 12, Dezember 2010, S. 4620–4622, doi:10.1128/JCM.02019-10. PMID 20962142. PMC 3008463 (freier Volltext).
- ↑ Oxoid™ VET-RPLA Toxin Detection Kit. In: thermofisher.com. Thermo Fisher Scientific, abgerufen am 9. Januar 2017 (englisch).
- ↑ R. J. Almeida, F. W. Hickman-Brenner u. a.: Comparison of a latex agglutination assay and an enzyme-linked immunosorbent assay for detecting cholera toxin. In: Journal of clinical microbiology. Band 28, Nummer 1, Januar 1990, S. 128–130, PMID 2298870. PMC 269552 (freier Volltext).
- ↑ L. J. Coupland, I. McElarney u. a.: Simultaneous detection of viral and bacterial enteric pathogens using the Seeplex® Diarrhea ACE detection system. In: Epidemiology and infection. [elektronische Veröffentlichung vor dem Druck] Dezember 2012, doi:10.1017/S0950268812002622. PMID 23211606.
- ↑ a b c d Michael T. Madigan, John M. Martinko, Jack Parker: Brock Mikrobiologie. Deutsche Übersetzung herausgegeben von Werner Goebel, 1. Auflage. Spektrum Akademischer Verlag GmbH, Heidelberg/Berlin 2000, ISBN 978-3-8274-0566-1, S. 1098–1099.
- ↑ Fallbericht: Nachweis von Vibrio cholerae non-O1, non-O139 bei einer immunsupprimierten Patientin nach Baden in Binnengewässern. In: Robert Koch-Institut (Hrsg.): Epidemiologisches Bulletin. Nr. 34/2006, 25. August 2006, S. 295.
- ↑ a b D. Dobosch, A. Gomez Zavaglia, A. Kuljich: The role of food in cholera transmission. In: Medicina. Band 55, Nummer 1, 1995, S. 28–32, PMID 7565031.
- ↑ a b c d Oliver Liesenfeld: Vibrionen, Aeromonas. In: Helmut Hahn, Stefan H. E. Kaufmann, Thomas F. Schulz, Sebastian Suerbaum (Hrsg.): Medizinische Mikrobiologie und Infektiologie. 6. Auflage. Springer Verlag, Heidelberg 2009, ISBN 978-3-540-46359-7, S. 269–274.
- ↑ NCBI: Vibrio cholerae O1 (serogroup)
- ↑ NCBI: Vibrio cholerae O139 (serogroup)
- ↑ NCBI: cholerae O1 biovar El Tor str. Inaba *&srchmode=2 str. Inaba (strains)
- ↑ NCBI: cholerae O1 biovar El Tor str. Ogawa *&srchmode=2 str. Ogawa (strains)
- ↑ a b A. K. Siddique, A. H. Baqui u. a.: Survival of classic cholera in Bangladesh. In: Lancet. Band 337, Nummer 8750, Mai 1991, S. 1125–1127, PMID 1674016.
- ↑ a b c d M. Li, M. Kotetishvili, Y. Chen, S. Sozhamannan: Comparative genomic analyses of the vibrio pathogenicity island and cholera toxin prophage regions in nonepidemic serogroup strains of Vibrio cholerae. In: Applied and environmental microbiology. Band 69, Nummer 3, März 2003, S. 1728–1738, PMID 12620865. PMC 150053 (freier Volltext).
- ↑ Michael T. Madigan, John M. Martinko, Jack Parker: Brock Mikrobiologie. Deutsche Übersetzung herausgegeben von Werner Goebel, 1. Auflage. Spektrum Akademischer Verlag GmbH, Heidelberg/Berlin 2000, ISBN 978-3-8274-0566-1, S. 95.
- ↑ S. M. McLeod, H. H. Kimsey, B. M. Davis, M. K. Waldor: CTXphi and Vibrio cholerae: exploring a newly recognized type of phage-host cell relationship. In: Molecular microbiology. Band 57, Nummer 2, Juli 2005, S. 347–356, doi:10.1111/j.1365-2958.2005.04676.x. PMID 15978069.
- ↑ E. F. Boyd, K. E. Moyer, L. Shi, M. K. Waldor: Infectious CTXPhi and the vibrio pathogenicity island prophage in Vibrio mimicus: evidence for recent horizontal transfer between V. mimicus and V. cholerae. In: Infection and immunity. Band 68, Nummer 3, März 2000, S. 1507–1513, PMID 10678967. PMC 97308 (freier Volltext).
- ↑ Michael T. Madigan, John M. Martinko, Jack Parker: Brock Mikrobiologie. Deutsche Übersetzung herausgegeben von Werner Goebel, 1. Auflage. Spektrum Akademischer Verlag GmbH, Heidelberg/Berlin 2000, ISBN 978-3-8274-0566-1, S. 288.
- ↑ S. M. Faruque, J. Zhu u. a.: Examination of diverse toxin-coregulated pilus-positive Vibrio cholerae strains fails to demonstrate evidence for Vibrio pathogenicity island phage. In: Infection and immunity. Band 71, Nummer 6, Juni 2003, S. 2993–2999, PMID 12761075. PMC 155729 (freier Volltext).
- ↑ TRBA 466: Einstufung von Prokaryonten (Bacteria und Archaea) in Risikogruppen. In: Website der Bundesanstalt für Arbeitsschutz und Arbeitsmedizin (BAuA). 25. April 2012, S. 244, abgerufen am 9. März 2013.
- ↑ J. G. Morris, Jr.: Non-O1 group Vibrio cholerae strains not associated with epidemic disease. In: I. Kaye Wachsmuth, Paul A. Blake, Orjan Olsvik (Hrsg.): Vibrio cholerae and cholera: Molecular to global perspectives. 1. Auflage. ASM Press, Washington 1994, ISBN 1-55581-067-5.
- ↑ Text des Infektionsschutzgesetzes (IfSG) bei juris. Abgerufen am 9. März 2013.
- ↑ A. S. Vicari, C. Ruiz-Matus u. a.: Development of a cholera vaccination policy on the island of hispaniola, 2010-2013. In: The American journal of tropical medicine and hygiene. Band 89, Nummer 4, Oktober 2013, S. 682–687, doi:10.4269/ajtmh.13-0200. PMID 24106195. PMC 3795098 (freier Volltext).
- ↑ Text der Trinkwasserverordnung (TrinkwV 2001) bei juris. Abgerufen am 24. März 2013.
- ↑ Fachgruppe Lebensmittelmikrobiologie und -hygiene, Arbeitsgruppe Mikrobiologische Richt- und Warnwerte der DGHM e. V.: Mikrobiologische Richt- und Warnwerte zur Beurteilung von Lebensmitteln (Stand Mai 2012), Kapitel 21 Richt- und Warnwerte für Seefische. (Nicht mehr online verfügbar.) In: Website der Deutschen Gesellschaft für Hygiene und Mikrobiologie (DGHM). Archiviert vom Original am 11. Februar 2013; abgerufen am 24. März 2013.
- ↑ Frequently asked questions and information for travelers. (PDF; 25 kB) In: Health topics: Cholera auf der Webseite der Weltgesundheitsorganisation (WHO). Abgerufen am 24. März 2013.
Weblinks
- Forschungsprojekt VibrioNet (auf Englisch). Abgerufen am 8. Dezember 2013.
- Pathogenicity Project – Vibrio cholerae. In: Pathogenicity Project – Homepage. Abgerufen am 8. Dezember 2013.
- Copepods and cholera in untreated water. In: National Science Foundation (NSF). Abgerufen am 27. Oktober 2013.
- 150 Gene durch Raubmord erbeutet, auf Wissenschaft.de vom 8. Oktober 2019
- Bundesinstitut für Risikobewertung (BfR): Fragen und Antworten zu Vibrionen
- Javier Campos, Eriel Martínez, Karen Marrero, Yussuan Silva, Boris L. Rodríguez, Edith Suzarte, Talena Ledón, Rafael Fando: Novel type of specialized transduction for CTX phi or its satellite phage RS1 mediated by filamentous phage VGJ phi in Vibrio cholerae, in: J Bacteriol 185(24), Dezember 2003, S, 7231–7240, doi:10.1128/jb.185.24.7231-7240.2003, PMID 14645284, PMC 296256 (freier Volltext) – über das Satellitenvirus RS1 von CTXphi und einen weiteren Phagen VGJphi (offiziell Vibrio virus VGJ, Fam. Inoviridae, Gattung Fibrovirus).
- N. I. Smirnov, S. P. Zadnova, A. V. Toporkov: Effects of the recombinant plasmid carrying the genes of cholera prophages CTX and RS1 on the expression of virulence and immunogenicity genes in the cholera pathogen, in: Mol Gen Mikrobiol Virusol (3), S. 3–8, 2005, PMID 16173391 (Artikel auf russisch) – ebenfalls auch über RS1