Escherichia-Virus Lambda
| Escherichia-Virus Lambda | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ||||||||||||||||||||
| Systematik | ||||||||||||||||||||
| ||||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||||
| ||||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||||
Lambdavirus lambda
| ||||||||||||||||||||
| Kurzbezeichnung | ||||||||||||||||||||
| λ | ||||||||||||||||||||
| Links | ||||||||||||||||||||
|
Das Escherichia-Virus Lambda (wissenschaftlich Lambdavirus lambda, mit Referenzstamm englisch Escherichia phage lambda alias Lambda-Phage, Bakteriophage Lambda, Enterobacteria-Phage Lambda oder Phage λ) ist ein Virus aus der Klasse Caudoviricetes, das das Bakterium Escherichia coli zum Wirt hat; und wird daher als Bakteriophage klassifiziert. Dieser Phage hat in der Geschichte der Virusforschung und molekularen Genetik wie auch bei gentechnologischen Verfahren eine große Bedeutung. Er wurde 1950 von Esther Lederberg entdeckt.[3]
Morphologie
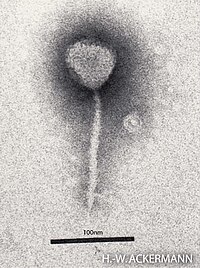
Das Virion des Lambda-Phagen ist wie bei allen Viren des Morphotyps der Siphoviren aus einem Kapsid mit der enthaltenden dsDNA und einem Schwanzteil aufgebaut. Das Kapsid hat eine isometrische, ikosaedrische Symmetrie und ist im Durchmesser etwa 60 nm groß; es besteht aus 72 Kapsomeren (60 Hexamere, 12 Pentamere, Triangulationszahl T=7). Aufgrund der Lücke für den Ansatz des Schwanzteils besteht das Kapsid jedoch nicht aus je 420 (= 60×6 + 12×5) Molekülen der beiden Haupt-Kapsidproteine E und D, sondern nur aus 415.
Der Schwanzteil ist flexibel, aber nicht-kontraktil und 8 × 150 nm groß. An dessen Ende befinden sich vier lange Fibern, die jedoch bei in in vitro gezüchteten Stämmen verschwinden.
Das lineare, grundsätzlich doppelsträngige DNA-Genom des Lambda-Phagen ist 48.502 bp (Basenpaare) groß und kodiert für etwa 70 Proteine. Die vollständige Genomsequenz wurde von Frederick Sanger 1982 ermittelt. Die lineare DNA besitzt an beiden Enden kurze einzelsträngige Abschnitte, die komplementär zueinander sind und der Rezirkularisierung der Phagen-DNA nach Injektion in den Wirt dienen. Diese Enden werden auch als kohäsive Enden (cos), homologe Bindeenden oder klebrige Enden (engl. sticky ends) bezeichnet.
Integration
Das Escherichia-Virus Lambda ist ein temperenter Bakteriophage. Seine lineare Phagen-DNA wird nach der Infektion der Wirtszelle zu einer zirkulären DNA geschlossen, indem an der cos-Stelle der DNA die sticky ends aus je 12 Basen von der DNA-Ligase des Bakteriums zusammengefügt werden.[4] Die zirkuläre DNA kann von der viralen Integrase in das Bakterienchromosom eingebaut werden. Die Integration der Phagen-DNA in das Wirtsgenom geschieht über die att-Stelle des Plasmids und ist eine ortsspezifische Rekombination und kann zu spezialisierter Transduktion führen. In diesem Zustand ist der Lambda-Phage ein Prophage und bleibt in das Wirtsgenom integriert, ohne seine Gene zu exprimieren.
Expression

Die Expression der Prophagen-Gene werden von einem Repressor-Protein unterdrückt. Das einzige Gen, das aktiv bleibt, ist das Repressorgen cI selbst. Das heißt, dass die Prophagen-DNA mit jeder Teilung der Wirtszelle ebenfalls dupliziert wird, aber seine Gene werden nicht transkribiert. Dieser Prozess ist auch als lysogener Zyklus bekannt. Die Beendigung dieser inaktiven Lysogenie kann durch Stressfaktoren wie z. B. Antibiotika, UV-Strahlung, Nährstoffmangel und insbesondere DNA-Schädigung induziert werden. Im letztgenannten Falle führt eine Schädigung der Erbinformation zur Aktivierung von RecA, einem Induktor von DNA-Reparaturprozessen, der SOS-Antwort. Jedoch spaltet RecA nach Aktivierung nicht nur sein eigentliches Substrat, sondern auch den Lambda-Repressor cI. Dieses kann nun nicht mehr dimerisieren, was zur Beendigung der Repression führt und damit auch zur Expression des Phagengenoms. Jetzt kann der Lambda-Phage in den lytischen Zyklus eintreten, bei dem die eigentliche Vermehrung des Phagen stattfindet. Nach Synthese der Phagenkomponenten und Zusammenbau erfolgt die Lyse der Zellen und damit die Freisetzung der reifen, infektiösen Phagen.
Anwendungen in der Gentechnik
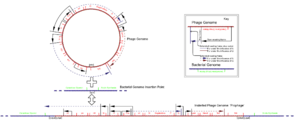
Lambda-Phagen werden in der Gentechnik als Insertions-Vektoren verwendet. Allerdings sind vor dem Einsatz als Vektor einige Modifikationen nötig. Zunächst sollten überzählige Restriktionsschnittstellen entfernt werden. Zur Einfügung eines zusätzlichen Genabschnitts (Insert) müssen ursprüngliche Phagen-Sequenzen entfernt werden, da sonst aufgrund des zu großen, künstlichen Genoms keine Verpackung in das Kapsid erfolgen kann. Dazu werden nicht zur Replikation benötigte Phagengene sowie die Gene für den lysogenen Zyklus entfernt. Dadurch entsteht Platz für ein ca. 8kb großes Insert. Escherichia-Virus Lambda wird sowohl als Klonierungsvektor für die Herstellung von cDNA-Genbanken verwendet, als auch als Plattform zum Phagen-Display.
Die isolierte DNA des Escherichia-Virus Lambda wird darüber hinaus als Größenmarker für Agarose-Gelelektrophorese eingesetzt. Durch den Verdau mit einer Restriktionsendonuklease entsteht ein Gemisch aus verschieden großen DNA-Fragmenten bekannter Größe, welche die Größenbestimmung von anderen DNA-Fragmenten nach ihrer Auftrennung erlauben. Häufig wird dabei das Restriktionsenzym PstI verwendet, da das hierbei entstehende Fragmentgemisch ein relativ breites Spektrum an Größen abdeckt.
Literatur
- Court, D.L. et al. (2007): A new look at bacteriophage lambda genetic networks. In: J. Bacteriol. Bd. 189, 298–304. PMID 17085553 doi:10.1128/JB.01215-06 PDF
- M. E. Gottesman, R. A. Weisberg (2004): Little lambda, who made thee? In: Microbiol. Mol. Biol. Rev. Bd. 68, S. 796–813. PMID 15590784 doi:10.1128/MMBR.68.4.796-813.2004 PDF
- C. Coleclough, F. L. Erlitz (1985): Use of primer-restriction-end adapters in a novel cDNA cloning strategy. In: Gene. Bd. 34, S. 305–314. PMID 2408965
- B. Hohn (1983): DNA sequences necessary for packaging of bacteriophage lambda DNA. In: Proc. Natl. Acad. Sci. U.S.A. Bd. 80, S. 7456–7460. PMID 6324174, PMC 389970 (freier Volltext)
- F. Sanger, A. R. Coulson, G. F. Hong, D. F. Hill, G. B. Petersen (1982): Nucleotide sequence of bacteriophage lambda DNA. In: J. Mol. Biol. Bd. 162, S. 729–773. PMID 6221115
Weblinks
Einzelnachweise
- ↑ ICTV: ICTV Master Species List 2021.v2, New MSL including some corrections.
- ↑ a b c ICTV: ICTV Taxonomy history: Enterobacteria phage T4, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- ↑ Invisible Esther jax.org
- ↑ Lambda-Phage. Abgerufen am 15. Januar 2020.