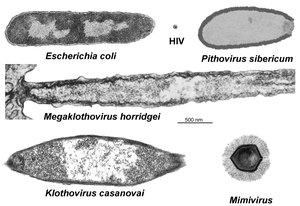
Riesenvirus
Die hier behandelte Gruppe ist als Taxon durch neue Forschungen obsolet geworden oder aus anderen Gründen nicht Teil der offiziellen Virus-Taxonomie.
Als Riesenvirus (englisch giant virus, GV, oder kurz
) werden sehr große Viren bezeichnet. Zwar variieren die genauen Kriterien in der wissenschaftlichen Literatur, im Allgemeinen werden als Riesenviren aber Viren mit einem großen Kapsid von mindestens 200 bis 400 nm bezeichnet, typischerweise umgeben von einer dicken Schicht Proteinfasern (Stärke ca. 100 nm). Damit sind viele Riesenviren größer als durchschnittliche Bakterien.[1][2]

Als Untergrenze für die Genomgröße werden meist 300 kbp (Kilobasenpaare; alle bekannten Riesenviren haben ein Doppelstrang-DNA-Genom) festgesetzt. Yutin und Koonin (2019) setzten allerdings eine etwas höhere Grenze von 500 kbp an,[3] ebenso Brandes und Linial (2019).[4]
Am oberen Ende wurden Genome mit 1000 kbp und mehr gefunden, etwa bei „Tupanvirus“ mit 1.516 kbp.
Das Genom der Riesenviren umfasst dabei eine Größenordnung von etwa 1000 kodierenden Genen (statt sonst kaum ein Dutzend). Dies ist extrem umfangreich im Vergleich zu anderen Virus-Genomen.[5][6][7]
Zur späten Entdeckung der meisten Riesenviren (zahlreich erst ab etwa 2003) trug bei, dass sie bei der Suche nach Viren in den Filtern (mit typischer Porengröße von 0,2 μm) hängen blieben, die Bakterien und Protisten von Viren abtrennen sollten, langsamer zu leicht sichtbaren Klumpen aggregieren und sich auch langsamer vermehren.[5]
Die bekannten Riesenviren gehören dem dsDNA-Virus-Phylum Nucleocytoviricota (veraltet
, NCLDV) an, und zwar den Klassen Megaviricetes (u. a. mit den Familien Mimiviridae und Phycodnaviridae) oder der Ordnung Asfuvirales aus der Klasse Pokkesviricetes (mit der Gattung Asfivirus, dem Erreger der Afrikanischen Schweinepest). Die Wirte der Riesenviren sind komplex-zelluläre Organismen (Eukaryonten).
Eine grafische Darstellung der Größenverhältnisse hat Laurie O'Keefe gegeben.[8]
Ähnlich wie Retroviren sind manche Vertreter der NCLDV in der Lage, sich in das Genom ihrer Wirtszellen zu integrieren (englisch endogenous viral element, EVE).[9]
Riesenphagen
Für große Viren von Prokaryonten (Bakterien oder Archaeen: Phagen) hat man eigene Größenkriterien geschaffen, da ihre Wirte naturgemäß nicht so viel Platz bieten. Auch hier sind alle bekannten Vertreter dsDNA-Viren. Näheres siehe Riesenphagen, Megaphagen und Jumbo-Phagen.
Einzelnachweise
- ↑ Kelly A. Reynolds: Mysterious Microbe in Water Challenges the Very Definition of a Virus. In: Water Conditioning & Purification. 2010. (via Web-Archi vom 19. März 2014)
- ↑ Ogata H, Toyoda K, Tomaru Y, Nakayama N, Shirai Y, Claverie JM, Nagasaki K: Remarkable sequence similarity between the dinoflagellate-infecting marine girus and the terrestrial pathogen African swine fever virus. In: Virology Journal. 6, Nr. 178, 2009–2010, S. 178. doi:10.1186/1743-422X-6-178. PMID 19860921. PMC 2777158 (freier Volltext).
- ↑ Eugene V. Koonin, Natalya Yutin: Evolution of the Large Nucleocytoplasmatic DNA Viruses of Eukaryotes and Convergent Origins of Viral Gigantism, in: Advances in Virus research, Band 103, AP 21. Januar 2019, doi:10.1016/bs.aivir.2018.09.002, S. 167–202. Die Klosneuviren sind teilweise als Klosneviren fehlgeschrieben.
- ↑ Nadav Brandes, Michal Linial: Giant Viruses – Big Surprises, in: Viruses 11(5); 404 vom 30. April 2019, doi:10.3390/v11050404, PMC 6563228 (freier Volltext), PMID 31052218
- ↑ a b James L. Van Etten, Leslie C. Lane, David D. Dunigan: DNA Viruses: The Really Big Ones (Giruses). Annu. Rev. Microbiol. 64, 2010, S. 83–99, doi:10.1146/annurev.micro.112408.134338 (Volltext frei zum persönlichen Gebrauch).
- ↑ James L. Van Etten: Giant Viruses. In: American Scientist. 99, Nr. 4, Juli–August 2011, S. 304–311. doi:10.1511/2011.91.304. (via Web-Archiv vom 11. Juni 2011)
- ↑ Legendre M, Arslan D, Abergel C, Claverie JM: Genomics of Megavirus and the elusive fourth domain of Life. In: Communicative & Integrative Biology. 5, Nr. 1, Januar 2012, S. 102–106. doi:10.4161/cib.18624. PMID 22482024. PMC 3291303 (freier Volltext).
- ↑ Laurie O'Keefe: Sizing Up Viruses, auf: The Scientist. Illustration zu Didier Raoult: Viruses Reconsidered, ebenda vom 28. Februar 2014
- ↑ Mohammad Moniruzzaman, Alaina R. Weinheimer, Carolina A. Martinez-Gutierrez, Frank O. Aylward: Widespread endogenization of giant viruses shapes genomes of green algae, in: nature vom 18. November 2020, doi:10.1038/s41586-020-2924-2, dazu:
Kendall Daniels: Lurking in genomic shadows: How giant viruses fuel the evolution of algae, vtnews, SciTechDaily, Quelle: Virginia Tech, 18. November 2020
Weblinks
- Jason R. Schrad, Jônatas S. Abrahão, Juliana R. Cortines and Kristin N. Parent: Structural and Proteomic Characterization of the Initiation of Giant Virus Infection, in: Cell vom 8. Mai 2020, doi:10.1016/j.cell.2020.04.032
- Jason R. Schrad, Jônatas S. Abrahão, Juliana R. Cortines, Kristin N. Parent: Boiling Acid Mimics Intracellular Giant Virus Genome Release, auf: bioRxiv vom 20. September 2019, doi:10.1101/777854 (Preprint)
- Mysterious Giant Viruses: Gargantuan in Size and Complexity, auf: SciTechDaily vom 9. Mai 2020, Quelle: Michigan State University
- Adrian De Novato: New study shines light on mysterious giant viruses, auf: phys.org vom 8. Mai 2020; die Abbildung zeigt offenbar einen typischen Vertreter der Gattung Mimivirus mit Stargate (Öffnung für DNA) und Tegument (Hülle aus Fibrillen).
- Masaharu Takemura: Medusavirus Ancestor in a Proto-Eukaryotic Cell: Updating the Hypothesis for the Viral Origin of the Nucleus, in: Front. Microbiol. 11:571831, 3. September 2020, doi:10.3389/fmicb.2020.571831
- Nicoletta Lanese: Giant viruses spew their DNA through a 'stargate.' Now, scientists know what triggers them, auf: LiveScience vom 26. Mai 2020
- GiantVirus.org: The largest known viral genomes (completely sequenced, > 170 kB) – Klasse Caudoviricetes fälschlicherweise als Familie Caudoviridae gelistet.
- Viral Dark Matter: Giant Viruses Have Metabolic Genes – Even Though Viruses Don’t Have a Metabolism. Auf: SciTechDaily vom 6. April 2020. Quelle: Virginia Tech.